 Estrutura de um ADN
Estrutura de um ADN
 Animacao de um ADN de dupla helice
Animacao de um ADN de dupla helice
O
acido desoxirribonucleico
(
ADN
, em portugues:
a
cido
d
esoxirribo
n
ucleico
[
nota 1
]
; ou
DNA
, em ingles:
d
eoxyribo
n
ucleic
a
cid
) e um
polimero
composto por duas cadeias
polinucleotidicas
que se enrolam umas sobre as outras para formar uma
dupla helice
. O polimero carrega instrucoes
geneticas
para o desenvolvimento, funcionamento, crescimento e reproducao de todos os
organismos
conhecidos e muitos
virus
. O ADN e o
acido ribonucleico
(RNA) sao
acidos nucleicos
. Ao lado de
proteinas
,
lipidios
e
carboidratos
complexos (
polissacarideos
), os acidos nucleicos sao um dos quatro principais tipos de
macromoleculas
essenciais para todas as formas de
vida
conhecidas.
E um
composto
organico
cujas
moleculas
contem as instrucoes
geneticas
que coordenam o desenvolvimento e funcionamento de todos os seres
vivos
e alguns
virus
, e que transmitem as caracteristicas
hereditarias
de cada ser vivo. A sua principal funcao e armazenar as informacoes necessarias para a construcao das
proteinas
de
ARNs
. Os segmentos de ADN que contem a
informacao genetica
sao denominados
genes
. O restante da sequencia de ADN tem importancia estrutural ou esta envolvido na regulacao do uso da informacao genetica.
A estrutura da molecula de ADN foi originalmente descoberta por
Rosalind Franklin
. No entanto, o
Premio Nobel de Fisiologia ou Medicina de 1962
foi entregue ao
norte-americano
James Watson
e ao
britanico
Francis Crick
, que se inspiraram em
Franklin
e demonstraram o funcionamento e a estrutura em dupla helice do ADN em 7 de Marco de 1953, juntamente com
Maurice Wilkins
.
Do ponto de vista quimico, o ADN e um longo
polimero
de unidades simples (
monomeros
) de
nucleotideos
, cuja cadeia principal e formada por moleculas de
acucares
e
fosfato
intercalados unidos por
ligacoes fosfodiester
. Ligada a molecula de acucar esta uma de quatro
bases nitrogenadas
mantidas juntas por
forcas hidrofobicas
.
[
2
]
A sequencia de bases ao longo da molecula de ADN constitui a informacao genetica. A leitura destas sequencias e feita por intermedio do
codigo genetico
, que especifica a sequencia linear dos
aminoacidos
das proteinas. A traducao e feita por um
ARN mensageiro
que copia parte da cadeia de ADN por um processo chamado
transcricao
e posteriormente a informacao contida neste e "traduzida" em proteinas pela
traducao
. Embora a maioria do ARN produzido seja usado na sintese de proteinas, algum ARN tem funcao estrutural, como por exemplo o
ARN ribossomico
, que faz parte da constituicao dos
ribossomos
.
Dentro da
celula
, o ADN pode ser observado numa estrutura chamada
cromossoma
durante a
metafase
. O conjunto de cromossomas de uma celula forma o
cariotipo
. Antes da
divisao celular
os cromossomas sao duplicados por meio de um processo chamado
replicacao
.
Eucariontes
como
animais
,
plantas
,
fungos
e
protozoarios
tem o seu ADN dentro do
nucleo
enquanto
procariontes
como as
bacterias
o tem disperso no
citoplasma
. Dentro dos cromossomas, proteinas da
cromatina
como as
histonas
compactam e organizam o ADN. Estas estruturas compactas guiam as interaccoes entre o ADN e outras proteinas, ajudando a controlar que partes do ADN sao transcritas.
 Estrutura quimica do ADN
Estrutura quimica do ADN
O ADN e um longo
polimero
formado por unidades repetidas chamadas
nucleotideos
.
[
3
]
[
4
]
A cadeia de ADN tem 2,2 a 2,4
nanometros
de largura, e um nucleotideo possui aproximadamente 0,33 nanometros de comprimento.
[
5
]
Embora os monomeros (nucleotideos) que constituem o ADN sejam muito pequenos, os polimeros de ADN podem ser moleculas enormes, com milhoes de nucleotideos. Por exemplo, o maior
cromossomo
humano (
cromossomo 1
), possui 220 milhoes de
pares de bases
de comprimento.
[
6
]
Uma molecula de ADN do ser humano possui aproximadamente dois metros de comprimento, encapsulada em um nucleo celular de 6 μm, o equivalente a acomodar uma linha de 40 km de comprimento em uma bola de tenis.
[
3
]
Em organismos vivos, o ADN nao existe como uma molecula unica (cadeia simples), mas sim como um par de moleculas firmemente associadas.
[
7
]
[
8
]
As duas longas cadeias de ADN enrolam-se como uma trepadeira formando uma
dupla helice
. Os nucleotideos estao presentes em ambas as cadeias da dupla helice, unidos com nucleotidos da mesma cadeia por ligacoes fosfodiester e a cadeia complementar por meio de pontes de hidrogenio formadas pelas suas bases. Em geral, uma base ligada a um acucar e chamada
nucleosideo
e uma base ligada a um acucar e um ou mais fosfatos e chamada nucleotideo. Portanto, o ADN pode ser referido como um
polinucleotideo
.
[
9
]
 Uma cadeia de ADN-B
Uma cadeia de ADN-B
A cadeia principal do ADN e formada por
fosfato
e residuos de
acucar
, dispostos alternadamente. O acucar no ADN e 2-desoxirribose, uma
pentose
(acucar com cinco
carbonos
). Os acucares sao unidos por
grupos fosfato
que formam
ligacoes fosfodiester
entre o terceiro e quinto atomos de carbono dos aneis de acucar adjacentes. Estas ligacoes assimetricas significam que uma cadeia de ADN tem uma direcao. Numa dupla helice, a direcao dos nucleotideos de uma cadeia e oposta a direcao dos nucleotideos da outra cadeia. O formato das cadeia do ADN e designado antiparalelo. As terminacoes assimetricas das cadeias de ADN sao designadas terminais
5'
(cinco linha) e
3'
(tres linha). Uma das diferencas principais entre o ADN e o ARN encontra-se no acucar, com a substituicao da 2-desoxirribose no ADN pela
ribose
no ARN.
[
3
]
A dupla helice do ADN e estabilizada por
pontes de hidrogenio
entre as bases presas as duas cadeias. As quatro bases encontradas no ADN sao a
adenina
(A),
citosina
(C),
guanina
(G) e
timina
(T). Estas quatro bases ligam-se ao acucar/fosfato para formar o nucleotideo completo.
[
3
]
Estas bases sao classificadas em dois tipos; a adenina e guanina sao
compostos heterociclicos
chamados
purinas
, enquanto a citosina e timina sao
pirimidinas
. Uma quinta base (uma pirimidina) chamada
uracila
(U) aparece no ARN e substitui a timina, a uracila difere da timina pela falta de um grupo de
metila
no seu anel. A uracila normalmente nao esta presente no ADN, so ocorrendo como um produto da decomposicao da citosina.
[
3
]
Excecoes para esta regra sao os
fagos
AR9, 3NT, I10, bem como o PBS1 (muito utilizado em pesquisas), que contem uracila no seu ADN, em vez de timina.
[
10
]
No topo, pareamento GC com tres pontes de hidrogenio. Em baixo, AT com duas pontes de hidrogenio.
Cada tipo de base numa cadeia forma uma ligacao com apenas um tipo de base na outra cadeia. Este comportamento e designado de
complementariedade de bases
. Assim, as purinas formam pontes de hidrogenio com pirimidinas, i.e. A liga-se com T e C com G. Este arranjo de dois nucleotideos complementares na dupla helice e chamado
par de bases
. Alem das pontes de hidrogenio entre as bases, as duas cadeias sao mantidas juntas devido a forcas geradas por
interacoes hidrofobicas
entre as bases
empilhadas
, a qual nao e influenciada pela sequencia do ADN.
[
11
]
Como as pontes de hidrogenio nao sao
ligacoes covalentes
, podem ser quebradas e reunidas com relativa facilidade. Desta forma, as duas fitas da dupla helice de ADN podem ser separadas como um
ziper
(fecho de correr) por forca mecanica ou altas temperaturas.
[
12
]
Como resultado desta complementariedade, toda a informacao contida numa das cadeias de ADN esta tambem contida na outra, o que e fundamental para a replicacao do ADN.
[
3
]
Os dois tipos de pares de base formam diferentes numeros de pontes de hidrogenio: AT forma duas pontes de hidrogenio enquanto GC formam tres pontes de hidrogenio. Desta forma a interacao entre GC e mais forte que AT. Como resultado, a percentagem de GC numa dupla fita de ADN determina a forca de interacao entre as duas cadeias.
[
13
]
Uma parte da dupla cadeia de ADN que precisa de ser separada facilmente, tal como a TATAAT
Caixa de Pribnow
nos
promotores
bacterianos, tende a ter sequencias com maior predominio de AT, para facilitar a abertura da dupla cadeia aquando da transcricao. No laboratorio, a forca desta interaccao pode ser medida encontrando a temperatura necessaria para quebrar as pontes de hidrogenio, a
temperatura de desnaturacao
(tambem chamado
T
m
). Quando todos os pares de base numa dupla helice de ADN quebram as suas ligacoes, as duas cadeias separam-se e existem em solucao como duas moleculas completamente independentes. Estas moleculas de ADN de cadeia simples nao tem uma unica forma comum, mas algumas conformacoes sao mais estaveis do que outras.
[
14
]
O ADN normalmente encontra-se em forma de uma espiral dextrogira (gira para a direita, ou no
sentido horario
). Portanto, as duas cadeias de nucleotideos giram uma sobre a outra e acabam por formar sulcos entre as cadeias de fosfato, deixando expostas as faces das bases nitrogenadas que nao estao unidas por pontes de hidrogenio com a base complementar.
[
15
]
Ha dois tipos de sulcos na superficie da dupla helice: um com 22 A denominado sulco maior e um com 12 A designado de sulco menor.
[
16
]
A principal funcao dos sulcos do ADN e fornecer a informacao acerca das bases que se encontram ligadas numa determinada regiao da dupla cadeia sem necessidade de abertura. O sulco maior oferece maior acessibilidade para ligacao com proteinas do que o sulco menor. Um exemplo disto e a
TBP
(
TATA-binding protein
) uma importante proteina para a transcricao em eucariotas.
[
17
]
Uma sequencia de ADN e chamada de
senso
se possui a mesma sequencia do
ARNm
. A cadeia oposta (complementar) a cadeia "senso" e denominada sequencia
antissenso
. Como a
ARN polimerase
sintetiza um ARN que e complementar a fita molde, entao podemos dizer que ela utiliza a cadeia anti-senso como molde para produzir um ARN. As sequencias senso e anti-senso podem existir em diferentes partes da mesma cadeia de ADN, que pode ser de um lado ou do outro, dependendo de onde se encontra a sequencia codificadora.
As vezes nao e possivel dizer qual e a cadeia senso ou antissenso. Isto acontece devido a existencia de genes que se sobrepoem. Neste caso ambas as cadeias dao origem a um ARN.
[
18
]
Nas
bacterias
, a sobreposicao pode estar envolvida da regulacao da transcricao.
[
19
]
Nos virus, a sobreposicao aumenta a capacidade do armazenamento de informacoes em pequenos genomas virais.
[
20
]
O ADN pode ser torcido num processo denominado superenrolamento. No estado
relaxado
do ADN, uma fita normalmente da uma volta completa ao eixo da dupla helice a cada 10,4 pares de base, mas se o ADN esta torcido, as cadeias ficam mais ou menos enroladas.
[
21
]
Se o ADN esta torcido na direcao da helice, e denominado um
superenrolamento positivo
e as bases estao unidas mais firmemente. Ja o
superenrolamento negativo
refere-se a uma torcao na direcao oposta, resultando num
afrouxamento
das bases. Na natureza, o ADN apresenta um ligeiro superenrolamento negativo que e causado pela acao da
enzima
topoisomerase
.
[
22
]
Estas enzimas tambem sao necessarias para aliviar o estresse de torcao causado no ADN durante os processos de
transcricao
e
replicacao
.
[
23
]
 Da direita para a esquerda, a estrutura do ADN A, B e Z
Da direita para a esquerda, a estrutura do ADN A, B e Z
O ADN pode existir em muitas formacoes diferentes. As formacoes mais comuns sao:
ADN-A
, ADN-B, ADN-C, ADN-D,
[
24
]
ADN-E,
[
25
]
ADN-H,
[
26
]
ADN-L,
[
24
]
ADN-P,
[
27
]
e ADN-Z.
[
28
]
Porem, so as formacoes de ADN A, B e Z foram encontradas em sistemas biologicos naturais. A formacao que o ADN adopta depende de varios fatores da propria sequencia de ADN: a intensidade e direcao do superenrolamento, modificacoes quimicas das bases e a solucao na qual o ADN esta presente (ex.: concentracao de
metais
,
ioes
e
poliaminas
).
[
29
]
Das tres formacoes referidas, a forma “B” e a mais comum nas condicoes encontradas nas celulas.
[
30
]
A forma “A” corresponde a espiral dextra mais larga, com um sulco menor largo e superficial e um sulco maior estreito e profundo. A forma “A” ocorre sob condicoes nao fisiologicas em amostras de ADN desidratadas, enquanto na celula pode ser produzida por pareamento hibrido de ADN e ARN ou pelo complexo enzima-ADN.
[
31
]
[
32
]
Em segmentos de ADN onde as bases foram quimicamente modificadas por
metilacao
, o ADN pode sofrer uma grande modificacao na sua formacao e adoptar a forma ADN-Z. A cadeia gira sobre o eixo da dupla helice para a esquerda, o oposto da forma mais comum ? ADN-B.
[
33
]
Esta estrutura e rara e pode ser reconhecida por proteinas especificas de ligacao com o ADN-Z. Pode estar envolvida na regulacao da transcricao.
[
34
]
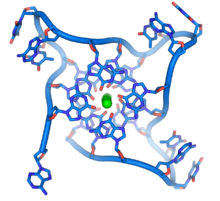 Estrutura de um quadruplex de ADN formado por repeticoes
telomericas
. A conformacao do esqueleto de ADN e diferente da tipica estrutura helicoidal.
[
35
]
Estrutura de um quadruplex de ADN formado por repeticoes
telomericas
. A conformacao do esqueleto de ADN e diferente da tipica estrutura helicoidal.
[
35
]
Nas extremidades do cromossomas lineares estao zonas especializadas do ADN chamadas
telomeros
. A funcao principal destas regioes e permitir que a celula replique as extremidades do cromossoma usando a enzima
telomerase
, porque enzimas que permitem replicar ADN normalmente nao conseguem copiar as extremidades 3' dos cromossomas.
[
36
]
Estas tampas de cromossoma especializadas tambem ajudam a proteger as extremidades do ADN, e evitam que o sistema de
reparacao de ADN
elimine estas regioes como erros que precisassem de ser corrigidos.
[
37
]
Em celulas humanas, os telomeros tem normalmente varios milhares de repeticoes de uma sequencia simples (TTAGGG).
[
38
]
Estas sequencias ricas em guanina podem estabilizar as extremidades dos cromossomas formando estruturas de unidades de quatro bases empilhadas, ao inves dos pares de base usuais encontrados em outras moleculas de ADN. Quatro bases de guanina formam uma placa chata e depois estas unidades chatas de quatro bases empilham-se no topo umas das outras, para formarem estruturas
quadruplex-G
estaveis.
[
39
]
Estas estruturas sao estabilizadas por
pontes de hidrogenio
entre as margens das bases e por
quelacao
de um iao metalico no centro de cada unidade de quatro bases.
[
40
]
Outras estruturas podem tambem ser formadas, com o conjunto central de quatro bases provenientes de uma cadeia simples enrolada a volta das bases ou de diversas cadeias paralelas, cada uma contribuindo com uma base para a estrutura central.
[
41
]
Alem destas estruturas empilhadas, os telomeros tambem formam grandes estruturas em forma de laco chamados
telomere loops
ou
T-loops
. O ADN de cadeia simples enrola-se a volta de um circulo grande estabilizado por proteinas que se ligam a telomeros.
[
42
]
Mesmo no fim dos
T-loops
, o ADN de cadeia simples do telomero e mantido sobre uma regiao de ADN de cadeia dupla pela cadeia do telomero que desestabiliza o ADN de dupla helice e o emparelhamento de bases de uma das duas cadeias. Esta estrutura de
cadeia tripla
e chamada de laco de deslocamento ou
D-loop
.
[
39
]
Estrutura da citosina com e sem o grupo 5-metil. Depois de desaminacao, a 5-metilcitosina tem a mesma estrutura da timina
A expressao de genes e influenciado pela maneira como o ADN esta disposto nos cromossomas, numa estrutura chamada
cromatina
. As modificacoes de bases podem estar envolvidas na disposicao, com as regioes quem tem expressao genica baixa ou inexistente contendo usualmente niveis elevados de
metilacao
de
citosina
. Por exemplo, a metilacao de citosina produz
5-metilcitosina
, que e importante na
inactivacao do cromossoma X
.
[
43
]
O nivel medio de metilacao varia entre organismos - o verme
Caenorhabditis elegans
tem pouca metilacao da citosina, enquanto
vertebrados
tem niveis mais elevados, com ate 1% do seu ADN contendo 5-metilcitosina
[
44
]
Apesar da importancia da 5-metilcitosina, esta pode
desaminar
transformando-se em timina. Citosinas metiladas sao por isso especialmente susceptiveis de sofrer
mutacoes
.
[
45
]
Outras modificacoes de bases incluem metilacao de adeninas em bacterias e
glicosilacao
do uracilo para produzir a "base-J" em organismos da classe
Kinetoplastida
.
[
46
]
[
47
]
 Benzopireno
, o maior mutagenio no
fumo do tabaco
, ligando-se ao ADN
[
48
]
Benzopireno
, o maior mutagenio no
fumo do tabaco
, ligando-se ao ADN
[
48
]
O ADN pode ser danificado por muitos tipos diferentes de
mutagenios
, que alteram a sequencia de ADN. Estes incluem
agentes oxidantes
,
agentes alquilantes
e tambem por
radiacao electromagnetica
de grande energia tal como luz
ultravioleta
e
raios-X
. O tipo de dano ao ADN produzido depende do tipo de mutagenio. A luz ultravioleta, por exemplo, pode danificar o ADN produzindo
dimeros de timina
, que sao ligacoes cruzadas entre pirimidinas.
[
49
]
Por outro lado, oxidantes como
radicais livres
ou
peroxido de hidrogenio
produzem multiplos tipos de danos, incluindo modificacoes de bases, em particular guanosina, e quebras das cadeias duplas.
[
50
]
Em cada celula humana, cerca de 500 bases podem sofrer danos por oxidacao por dia.
[
51
]
[
52
]
As quebras da cadeia dupla sao lesoes oxidativas de dificil reparacao, que podem produzir
mutacoes pontuais
,
insercoes
e
deleccoes
, assim como
translocacoes cromossomicas
.
[
53
]
Muitos mutagenios encaixam entre o espaco entre dois pares de bases adjacentes, na chamada
intercalacao
. A maioria dos intercaladores sao
aromaticos
e moleculas planas e incluem
brometo de etidio
,
daunomicina
,
doxorrubicina
e
talidomida
. Para que um intercalador encaixe entre pares de bases, as bases tem de se separar, abrindo a cadeia dupla. Isto inibe a transcricao e a replicacao do ADN, causando toxicidade e mutacoes. Como resultado, os intercaladores de ADN sao muitas vezes
carcinogenicos
.
Benzopireno
,
acridinas
,
aflatoxina
e brometo de etidio sao exemplos bem conhecidos.
[
54
]
[
55
]
[
56
]
No entanto, devido a sua capacidade de inibir a transcricao e replicacao, estas toxinas tambem sao usadas em
quimioterapia
para inibir o crescimento rapido de celulas tumorais.
[
57
]
O ADN ocorre normalmente como
cromossomas
lineares em eucariotas e como cromossomas circulares em procariotas. O conjunto dos cromossomas numa celula perfazem o seu
genoma
; o
genoma humano
tem aproximadamente 3 mil milhoes de pares de base dispostos em 46 cromossomas.
[
58
]
A informacao transportada pelo ADN esta contida nas
sequencias
de ADN chamados
genes
. A
transmissao
da informacao dos genes e conseguida pela complementaridade do emparelhamento das bases. Por exemplo, na transcricao, quando uma celula usa a informacao num gene, a sequencia de ADN e copiado para uma sequencia de ARN complementar por meio da atraccao entre o ADN e os nucleotideos de ARN correctos. Esta copia de ARN pode ser depois usada para compor uma sequencia proteica correspondente no processo de
traducao
, que depende da mesma interaccao entre nucleotideos de ARN. Alternativamente, uma celula pode simplesmente copiar a sua informacao genetica num processo chamado replicacao do ADN.
 T7 ARN polimerase
(azul) produzindo um ARNm (verde) a partir de um molde de ADN (laranja).
[
59
]
T7 ARN polimerase
(azul) produzindo um ARNm (verde) a partir de um molde de ADN (laranja).
[
59
]
O ADN genomico esta localizado no
nucleo celular
dos eucariontes, assim como em pequenas quantidades em
mitocondrias
e em
cloroplastos
. Em procariontes, o ADN esta dentro de um corpo de forma irregular no citoplasma chamado
nucleoide
.
[
60
]
A informacao genetica num genoma esta nos genes, e o conjunto completo desta informacao num organismo e chamado o seu
genotipo
. Um gene e a unidade basica da
hereditariedade
e e uma regiao do ADN que influencia uma caracteristica particular num organismo. Genes contem uma
fase aberta de leitura
que pode ser transcrita, assim como
sequencias reguladoras
tais como
promotores
ou
acentuassomos
, que controlam a transcricao da fase aberta de leitura.
Em muitas
especies
, apenas uma pequena fraccao da sequencia total do genoma codifica uma proteina. Por exemplo, apenas 1,5% do genoma humano consiste de
exoes
(que codificam proteinas), com mais de 50% do ADN humano consistindo de
sequencias repetitivas
.
[
61
]
As razoes para a presenca de tanto
ADN nao codificante
em genomas eucarioticos e as extraordinarias diferencas no
tamanho do genoma
, ou
valor C
, entre especies representam um enigma conhecido por
enigma do valor C
.
[
62
]
Contudo, sequencias de ADN que nao codificam proteinas podem ainda codificar moleculas de
ARN nao codificante
funcional, que estao envolvidas na regulacao da expressao genica.
[
63
]
Algumas sequencias de ADN nao codificante tem um papel estrutural nos cromossomas. Os
telomeros
e
centromeros
contem tipicamente poucos genes, mas sao importantes para a funcao e estabilidade dos cromossomas.
[
37
]
[
64
]
Uma forma abundante de ADN nao codificante em humanos sao os
pseudogenes
, que sao copias de genes que foram desabilitados por mutacao.
[
65
]
Estas sequencias sao usualmente apenas
fosseis
moleculares, apesar de poderem servir ocasionalmente como material genetico em bruto para a criacao de novos genes por meio do processo de
duplicacao de genes
e
divergencia
.
[
66
]
 Replicacao de ADN. A dupla helice e desdobrada por uma
helicase
e por uma
topoisomerase
. Em seguida, uma
ADN polimerase
produz uma copia da
cadeia lider
. Outra ADN polimerase liga-se a
cadeia atrasada
. Esta enzima produz segmentos descontinuos (chamados
fragmentos de Okazaki
) antes de a
ADN ligase
os juntar.
Replicacao de ADN. A dupla helice e desdobrada por uma
helicase
e por uma
topoisomerase
. Em seguida, uma
ADN polimerase
produz uma copia da
cadeia lider
. Outra ADN polimerase liga-se a
cadeia atrasada
. Esta enzima produz segmentos descontinuos (chamados
fragmentos de Okazaki
) antes de a
ADN ligase
os juntar.
Um gene e uma sequencia de ADN que contem informacao genetica e pode influenciar o
fenotipo
de um organismo. Dentro de um gene, a sequencia de bases ao longo de uma cadeia de ADN definem uma cadeia de
ARN mensageiro
, que por sua vez define uma ou mais sequencias proteicas. A relacao entre a sequencia de nucleotidos de um gene e a sequencia de
aminoacidos
de uma proteina e determinada pelas regras de
traducao
, conhecidas colectivamente como o
codigo genetico
. O codigo genetico consiste de 'palavras' de tres letras chamadas
codoes
formadas por uma sequencia de tres nucleotidos (p.e. ACU, CAG, UUU).
[
67
]
Na transcricao, os codoes de um gene sao copiados para um ARN mensageiro pela
ARN polimerase
. Esta copia de ARN e depois descodificada por um
ribossoma
que le a sequencia de ARN emparelhando o ARN mensageiro com o
ARN de transferencia
, que carrega aminoacidos. Uma vez que ha quatro bases em combinacoes de 3 letras, ha 64 codoes possiveis (
 combinacoes). Estas codificam os vinte aminoacidos, dando a maioria dos aminoacidos mais do que um codao possivel. Ha tambem tres codoes 'stop' ou 'nonsense' significando o fim da regiao codificante; estes sao os codoes UAA, UGA e UAG.
[
68
]
combinacoes). Estas codificam os vinte aminoacidos, dando a maioria dos aminoacidos mais do que um codao possivel. Ha tambem tres codoes 'stop' ou 'nonsense' significando o fim da regiao codificante; estes sao os codoes UAA, UGA e UAG.
[
68
]
A
divisao celular
e essencial para que um organismo cresca, mas quando uma celula se divide tem de replicar o ADN do seu genoma para que as duas celulas-filha tenham a mesma informacao genetica que a celula parental. A estrutura em dupla-helice do ADN fornece um mecanismo simples para a sua replicacao. As duas cadeias sao separadas e sequencias de ADN complementares a cada uma das cadeias sao recriadas por uma
enzima
chamada
ADN polimerase
. Esta enzima constroi a cadeia complementar encontrando a base correcta por intermedio do emparelhamento com a base complementar, e ligando-a a cadeia original. Como as polimerases de ADN so conseguem fazer a extensao de uma cadeia de ADN na direccao 5' para 3', outros mecanismos sao usados para copiar a cadeia antiparalela da dupla helice.
[
69
]
Desta forma, a base presente na cadeia antiga determina que base vai aparecer na nova cadeia e a celula acaba com uma copia perfeita do seu ADN.
Todas as funcoes do ADN dependem de interaccoes com proteinas. Estas interaccoes com proteinas podem ser nao especificas, ou a proteina pode ligar-se especificamente a uma unica sequencia de ADN. Algumas enzimas tambem se podem ligar ao ADN. Destas, as polimerases que copiam as sequencias de ADN na transcricao e replicacao sao particularmente importantes.
Proteinas que se ligam ao ADN (
DNA-binding
)
[
editar
|
editar codigo-fonte
]
Interaccao do ADN com
histonas
(mostrado em branco, em cima). Os aminoacidos basicos destas proteinas (em baixo a esquerda, em azul) liga-se aos grupos fosfato do ADN (em baixo a direita, em vermelho).
Proteinas estruturais que se ligam ao ADN sao exemplos bem estudados de interaccoes nao especificas ADN-proteinas. Nos cromossomas, o ADN esta ligado a proteinas estruturais formando complexos. Estas proteinas organizam o ADN numa estrutura compacta, a
cromatina
. Em eucariontes esta estrutura envolve a ligacao do ADN a um complexo de pequenas proteinas basicas chamadas histonas, enquanto em procariontes estao envolvidas varios tipos de proteinas.
[
70
]
[
71
]
As histonas formam um complexo em forma de disco, o
nucleossoma
, que contem duas voltas completas de ADN de cadeia dupla a sua volta. Estas interaccoes nao especificas formam-se quando os residuos basicos das histonas fazem
ligacoes ionicas
ao esqueleto acucar-fosfato acidico do ADN, e por isso sao largamente independentes da sequencia de bases.
[
72
]
Modificacoes quimicas nestes residuos de amino-acidos incluem
metilacao
,
fosforilacao
e
acetilacao
.
[
73
]
Estas mudancas quimicas alteram a forca da interaccao entre o ADN e as histonas, tornando o ADN mais ou menos acessivel a
factores de transcricao
e mudando a taxa de transcricao.
[
74
]
Outras proteinas com ligacao a ADN nao especificas incluem o grupo de proteinas de alta mobilidade, que se ligam a ADN dobrado ou distorcido.
[
75
]
Estas proteinas sao importantes pois dobram conjuntos de nucleossomas e organizam-nos em estruturas maiores que constituem os cromossomas.
[
76
]
Um grupo distinto destas proteinas sao as que se ligam especificamente a ADN de cadeia simples. Nos humanos, a proteina de replicacao A e o membro desta familia mais bem compreendido e e usado em processos onde a dupla helice e separada, incluindo durante a replicacao do ADN, recombinacao e reparo.
[
77
]
Estas proteinas parecem estabilizar ADN de cadeia dupla e protegem-no da formacao de
hairpin loops
e da degradacao por
nucleases
.
 O factor de transcricao do
helice-volta-helice
lambda repressor ligado ao seu alvo de ADN.
[
78
]
O factor de transcricao do
helice-volta-helice
lambda repressor ligado ao seu alvo de ADN.
[
78
]
Em contraste, outras proteinas evoluiram de modo a ligar-se a sequencias de ADN especificas. Os
factores de transcricao
sao dos mais intensivamente estudados (proteinas que regulam a transcricao). Cada factor de transcricao liga-se a um conjunto particular de sequencias de ADN e activa ou inibe a transcricao de genes que tenham estas sequencias perto dos seus promotores. Os factores de transcricao fazem isto de duas maneiras. Primeiro, podem ligar-se a polimerase do ARN responsavel pela transcricao, quer directamente quer por meio de proteinas mediadoras; isto posiciona a polimerase no promotor e permite que comece a transcricao.
[
79
]
Em alternativa, os factores de transcricao podem ligar-se a
enzimas
que modificam as histonas no promotor; isto muda a acessibilidade do molde de ADN a polimerase.
[
80
]
Como estes locais de ligacao podem ocorrer pelo genoma inteiro de um organismo, mudancas na actividade de um tipo de factor de transcricao pode afectar milhares de genes.
[
81
]
Por consequencia, estas proteinas sao muitas vezes alvo de processos de
transducao de sinal
que controlam respostas a mudancas ambientais ou diferenciacao e desenvolvimento celular. A especificidade da interaccao destes factores de transcricao com o ADN provem das proteinas que fazem contactos multiplos com a extremidade das bases de ADN, permitindo a leitura da sequencia de ADN. A maior parte destas interaccoes com bases faz-se no sulco maior, onde as bases estao mais acessiveis.
[
82
]
 A
enzima de restricao
EcoRV
(verde) num complexo com o seu ADN substrato.
[
83
]
A
enzima de restricao
EcoRV
(verde) num complexo com o seu ADN substrato.
[
83
]
As
nucleases
sao
enzimas
que cortam as cadeias de ADN mediante a
catalise
da
hidrolise
das
ligacoes fosfodiester
. As nucleases que hidrolisam
nucleotidos
a partir dos extremos das cadeias de ADN denominam-se
exonucleases
, enquanto as
endonucleases
cortam no interior das cadeias. As nucleases que se utilizam com maior frequencia em
biologia molecular
sao as
enzimas de restricao
, endonucleases que cortam o ADN em sequencias especificas. Por exemplo, a enzima EcoRV, mostrada a esquerda, reconhece a sequencia de 6 bases 5′-GAT|ATC-3′ e faz um corte em ambas as cadeias na linha vertical indicada, gerando duas moleculas de ADN. Outras enzimas de restricao geram, no entanto, extremidades coesivas, ja que cortam de forma diferente as duas cadeias de ADN. Na natureza, estas enzimas protegem as
bacterias
contra as infeccoes de
fagos
, ao digerir o ADN do fago quando entra atraves da parede bacteriana, actuando como um mecanismo de defesa.
[
84
]
Em
biotecnologia
, estas nucleases especificas utilizam-se na
clonagem molecular
e na tecnica de
impressao de ADN
(
fingerprinting
, em ingles).
As enzimas denominadas
ADN ligases
podem reunir pedacos de ADN cortados ou quebrados.
[
85
]
As ligases sao particularmente importantes na
replicacao do ADN
da cadeia atrasada de ADN, ja que unem os fragmentos curtos de ADN gerados no garfo de replicacao para formar uma copia completa do molde de ADN. Tambem se utilizam no
reparo de ADN
e na
recombinacao genetica
.
[
85
]
As
topoisomerases
sao enzimas que possuem actividade de nuclease e ligase. Estas proteinas mudam a quantidade de
ADN superenrolado
. Algumas destas enzimas funcionam cortando a helice de ADN e permitindo a uma seccao que faca rotacao, de maneira a reduzir o grau de superenrolamento; uma vez feito isto, a enzima volta a unir os fragmentos de ADN.
[
22
]
Outros tipos de enzimas sao capazes de cortar uma helice de ADN e depois passar a segunda cadeia de ADN atraves desta quebra, antes de reunir as helices.
[
86
]
As topoisomerases sao necessarias para muitos processos em que intervem o ADN, como a
replicacao
e a
transcricao
.
[
23
]
As
helicases
sao proteinas que pertencem ao grupo dos
motores moleculares
. Utilizam energia quimica armazenada nos trifosfatos de nucleosidos, fundamentalmente
ATP
, para romper pontes de hidrogenio entre bases e separar a dupla helice de ADN em cadeias simples. Estas enzimas sao essenciais para a maioria dos processos em que as enzimas necessitam de aceder as bases do ADN.
[
87
]
As
polimerases
sao
enzimas
que sintetizam cadeias de nucleotidos a partir de trifosfatos de nucleosidos. A sequencia de seus produtos sao copias de cadeias de polinucleotidos existentes, que se denominam
moldes
. Estas enzimas funcionam adicionando nucleotidos ao grupo
hidroxilo
em 3' do nucleotido anterior numa cadeia de ADN. Por consequencia, todas as polimerases funcionam na direccao 5′ → 3′.
[
88
]
Nos
sitios activos
destas enzimas, o trifosfato de nucleosido que se incorpora emparelha a sua base com a correspondente no molde: isto permite que a polimerase sintetize de forma precisa a cadeia complementar ao molde.
As polimerases classificam-se de acordo com o tipo de molde que utilizam:
- Na
replicacao do ADN
, uma
ADN polimerase dependente de ADN
realiza uma copia de ADN a partir de uma sequencia de ADN. A precisao e vital neste processo, por isso muitas destas polimerases possuem uma actividade de verificacao de leitura (
proofreading
). Mediante esta actividade, a polimerase reconhece erros ocasionais na reaccao de sintese, devido a falta de emparelhamento entre o nucleotido erroneo e o molde, o que gera um desacoplamento (
mismatch
). Se se detecta um desacoplamento, activa-se uma actividade
exonuclease
na direccao 3′ → 5′ e a base incorrecta e eliminada.
[
89
]
Na maioria dos organismos, as ADN polimerases funcionam num grande complexo denominado
replissoma
, que contem multiplas unidades acessorias, como
helicases
.
[
90
]
- As
ADN polimerases dependentes de ARN
sao uma classe especializada de polimerases que copiam a sequencia de uma cadeia de ARN em ADN. Incluem a
transcriptase reversa
, que e uma enzima
viral
implicada na infeccao de celulas por
retrovirus
, e a
telomerase
, que e necessaria para a replicacao dos telomeros.
[
36
]
[
91
]
A telomerase e uma polimerase inusual, porque contem o seu proprio molde de ARN como parte da sua estrutura.
[
37
]
- A
transcricao
e levada a cabo por uma
ARN polimerase dependente de ADN
que copia a sequencia de uma das cadeias de ADN em ARN. Para comecar a transcrever um gene, a ARN polimerase une-se a uma sequencia do ADN denominada
promotor
, e separa as cadeias de ADN. Entao copia a sequencia do gene num transcrito de
ARN mensageiro
ate que alcanca uma regiao do ADN denominada
terminador
, onde se detem e se separa do ADN. Como ocorre com as ADN polimerases dependentes de ADN em humanos, a ARN polimerase II (a enzima que transcreve a maioria dos genes do genoma humano) funciona como um grande complexo multiproteico que contem multiplas subunidades reguladoras e acessorias.
[
92
]
 A recombinacao implica a rotura e reuniao de dois (M e F) para produzir dois cromossomas novos, reorganizados (C1 e C2)
A recombinacao implica a rotura e reuniao de dois (M e F) para produzir dois cromossomas novos, reorganizados (C1 e C2)
Uma helice de ADN normalmente nao interage com outros segmentos de ADN. Nas celulas humanas os diferentes cromossomas ocupam areas separadas no
nucleo celular
denominadas “territorios cromossomicos”.
[
94
]
A separacao fisica dos diferentes cromossomas e importante para que o ADN mantenha a sua capacidade de funcionar como um armazem estavel de informacao. Um dos poucos momentos em que os cromossomas interagem e durante o
sobrecruzamento cromossomico
(
chromosomal crossover
, em ingles), durante o qual se
recombinam
. O sobrecruzamento cromossomico ocorre quando duas helices de ADN se rompem, sofrem intercambio e se unem novamente.
[
95
]
A recombinacao permite aos cromossomas trocar informacao genetica e produzir novas combinacoes de genes, o que aumenta a eficiencia da
seleccao natural
e pode ser importante na evolucao rapida de novas proteinas.
[
96
]
Durante a profase I da
meiose
, uma vez que os cromossomas homologos estao perfeitamente emparelhados formando estruturas que se denominam bivalentes, produz-se o fenomeno de sobrecruzamento ou entrecruzamento
(crossing-over)
, no qual os cromatideos homologos nao irmaos (procedentes do pai e da mae) trocam material genetico. A recombinacao genetica resultante faz aumentar em grande medida a variacao genetica entre a descendencia de progenitores que se reproduzem por via sexual. A recombinacao genetica tambem pode estar implicada na
reparacao do ADN
, em particular na resposta celular as roturas da dupla cadeia (
double-strand breaks
).
[
97
]
A forma mais frequente de sobrecruzamento cromossomico e a
recombinacao homologa
, na qual os dois cromossomas implicados compartilham sequencias muito similares. A recombinacao nao homologa pode ser danosa para as celulas, ja que pode produzir
translocacoes cromossomicas
e anormalidades geneticas. A reaccao de recombinacao e catalisada por enzimas conhecidas como
recombinases
, tais como a
RAD51
.
[
98
]
O primeiro passo no processo de recombinacao e uma rotura da dupla cadeia, causada por uma endo
nuclease
ou por dano no ADN.
[
99
]
Posteriormente, uma serie de passos catalisados em parte pela recombinase conduz a uniao das duas helices formando pelo menos uma
juncao de Holliday
, na qual um segmento de uma cadeia simples e anelada com a cadeia complementar na outra helice. A juncao de Holliday e uma estrutura de uniao tetraedrica que pode mover-se ao longo do par de cromossomas, intercambiando uma cadeia por outra. A reaccao de recombinacao detem-se pelo corte da uniao e a reuniao dos segmentos de ADN libertados.
[
100
]
O ADN contem a informacao genetica que permite a maioria dos organismos vivos funcionar, crescer e reproduzir-se. No entanto, desconhece-se o intervalo de tempo durante o qual ele exerceu esta funcao nos ~3000 milhoes de anos desde a
historia da vida
, ja que se propos que as formas de vida mais precoces poderiam ter utilizado
ARN
como material genetico.
[
101
]
[
102
]
O ARN poderia ter funcionado como parte central de um metabolismo primordial, ja que pode transmitir informacao genetica e simultaneamente actuar como
catalisador
, formando parte das
ribozimas
.
[
103
]
Este antigo
mundo de ARN
onde os acidos nucleicos funcionariam como catalisadores e como armazens de informacao genetica, poderia ter influenciado na
evolucao
do
codigo genetico
actual, baseado em quatro
nucleotidos
. Isto se deveria a que o numero de bases unicas num organismo e determinado entre um numero pequeno de bases (o que aumentaria a precisao da replicacao) e um numero grande de bases (que por sua vez aumentaria a eficiencia catalitica das ribozimas).
[
104
]
Infelizmente, nao dispomos de evidencia directa dos sistemas geneticos ancestrais, porque a recuperacao do ADN a partir da maior parte dos fosseis e impossivel. O ADN e capaz de sobreviver no meio ambiente durante menos de um milhao de anos, e logo comeca a degradar-se lentamente em fragmentos de menor tamanho em solucao.
[
105
]
Algumas investigacoes pretendem a obtencao de ADN mais antigo, como no caso do isolamento de uma bacteria viavel a partir de um cristal salino de 250 milhoes de anos de antiguidade,
[
106
]
mas estes dados sao controversos.
[
107
]
[
108
]
No entanto, podem utilizar-se ferramentas de evolucao molecular para inferir os genomas de organismos ancestrais a partir de organismos contemporaneos.
[
109
]
[
110
]
Em muitos casos, estas inferencias sao suficientemente fiaveis, de maneira que uma biomolecula codificada num genoma ancestral pode ser ressuscitada no laboratorio para ser estudada hoje.
[
111
]
[
112
]
Uma vez recomposta a biomolecula ancestral, suas propriedades poderiam oferecer informacoes sobre os ambientes primordial, remetendo ao campo emergente da
paleogenetica experimental
.
[
113
]
Apesar de tudo, o processo de trabalho
retrospectivo
tem limitacoes inerentes, razao pela qual outros investigadores tentam elucidar o mecanismo evolutivo trabalhando desde a origem da Terra ate adiante no tempo. Dada suficiente informacao sobre como as substancias cosmicas poderiam haver-se depositado na Terra e sobre as transformacoes que poderiam ter tido lugar na superficie terrestre, talvez poderiamos ser capazes de desenvolver modelos prospectivos de evolucao da informacao genetica.
 Friedrich Miescher
.
Friedrich Miescher
.
A historia do ADN comeca no final da
decada de 1860
, com a chegada do bioquimico e medico suico
Friedrich Miescher
(
1844
-
1895
) a
Universidade de Tubingen
, uma pacata cidade no sul da
Alemanha
. O jovem pesquisador estava disposto a dedicar-se ao estudo da quimica da celula e escolheu essa universidade porque nela o quimico
Felix Hoppe-Seyler
(1825-1895) havia inaugurado um importante laboratorio de
quimica fisiologica
. Na epoca floresciam ideias a respeito das origens e funcoes das celulas, apos a queda da teoria da
geracao espontanea
. A teoria celular estabelecia-se como um dos pilares da Biologia. Por tudo isso, as celulas atraiam a atencao de estudantes entusiasmados, como Miescher.
[
114
]
Felix Hoppe-Seyler foi quem primeiro descreveu as interacoes entre a
hemoglobina
, a proteina responsavel pela cor do sangue, e o gas
oxigenio
. Seu trabalho levou-o a interessar-se pela composicao bioquimica dos
linfocitos
. Mas Miescher enfrentou dificuldades para obter amostras com linfocitos em quantidade e grau de pureza adequados. Por sugestao de Hoppe-Seyler, Miescher comecou a estudar a quimica das celulas do pus; o material para a pesquisa era abundante, pois dezenas de bandagens com material purulento eram diariamente descartadas por um hospital proximo a universidade. Miescher desenvolveu tecnicas adequadas para o isolamento das celulas presentes em pus das bandagens e para a sua analise quimica. O objetivo inicial era investigar as proteinas celulares, um grupo de substancias descoberto cerca de trinta anos antes.
[
114
]
Em um dos seus muitos experimentos com celulas do
pus
, Miescher obteve um precipitado que diferia quimicamente de todas as substancias proteicas conhecidas. Ele descobriu que a nova substancia concentrava-se no nucleo celular, na epoca considerado uma estrutura de pouca importancia para o funcionamento celular. Aprimorando os metodos de
extracao
e
purificacao
da nova substancia, Miescher pode realizar uma analise quimica mais precisa, que mostrou que as quantidades relativas de
hidrogenio
,
carbono
,
oxigenio
e
nitrogenio
presentes diferiam das encontradas em proteinas, alem de uma quantidade incomum de
fosforo
. A substancia descoberta Miescher denominou
nucleina
, pelo fato de ela estar concentrada no nucleo das celulas.
[
114
]
O trabalho sobre nucleina so foi publicado em
1871
, apos certa resistencia do editor da revista cientifica, o proprio Hoppe-Seyler, que, no inicio, nao acreditou nos resultados apresentados por Miescher. Mesmo depois da publicacao do trabalho, muitos pesquisadores continuaram duvidando da existencia da nucleina; na opiniao deles, o achado de Miescher devia ser uma mistura de fosfato inorganico e proteinas.
[
114
]
[
115
]
As desconfiancas quanto a real existencia da nova substancia descrita por Miescher so foram superadas por volta de 1889, quando
Richard Altmann
(1852-1900) obteve preparacoes altamente purificadas de nucleina, sem nenhuma contaminacao por proteinas. Pelo fato de a substancia ter carater acido, o que ja havia sido detectado por Miescher, Altmann sugeriu que ela fosse chamada de
acido nucleico
em vez de nucleina.
[
116
]
Outro pesquisador pioneiro na descoberta foi
Albrecht Kossel
(1853-1927). Em 1877, ele juntou-se ao grupo de pesquisa de Hoppe-Seyler, entao trabalhando na Universidade de Estrasburgo (Franca), e comecou a estudar a composicao quimica das nucleinas. Kossel detectou dois tipos de bases nitrogenadas ja conhecidas, a
adenina
e a
guanina
. Em 1893, identificou uma nova base nitrogenada, que era liberada pela degradacao de nucleina das celulas do
timo
; por isso denominou-a
timina
. Logo em seguida, descobriu que a nucleina continha um quarto tipo de base nitrogenada, a qual denominou
citosina
.
[
117
]
Em 1894, o grupo liderado por Kossel descobriu que os acidos nucleicos continham tambem
pentose
, um acucar com cinco atomos de carbono.
[
118
]
Em reconhecimento as suas contribuicoes na area, foi agraciado em
1910
com o
Nobel de Fisiologia ou Medicina
.
[
119
]
Em 1909,
Phoebus Levene
e
Walter Abraham Jacobs
(1883-1967) conseguiram determinar a organizacao das moleculas de fosfato, de pentose e base nitrogenada no acido nucleico.
[
120
]
Esses tres componentes estao unidos entre si formando uma unidade fundamental, o
nucleotideo
. Em 1929, Levene e colaboradores identificaram pentoses componente do acido nucleico das celulas do timo, que denominaram 2-deoxi-D-ribose, pelo fato de ela possuir, no carbono 2 de sua cadeia, um
atomo
de oxigenio a menos que a ribose, uma pentose ja conhecida, encontrada pelos pesquisadores em dois tipos de acidos nucleicos: o acido ribonucleico, ou ribose, e o acido desoxirribonucleico, ou ADN, cujo acucar e a desoxirribose.
[
121
]
[
122
]
 Frederick Griffith em 1936
Frederick Griffith em 1936
Frederick Griffith
fez uma importante observacao no curso dos experimentos com a
bacteria
Streptococcus pneumoniae
em 1928. Esta bacteria, que causa
pneumonia
em humanos, normalmente e letal em
camundongos
. Entretanto algumas linhagens desta especie de bacterias eram menos virulentas (menos capazes de causar doencas ou morte). Nos experimentos de Griffith, ele usou duas linhagens distinguiveis pelas suas colonias quando cultivadas em laboratorio. Uma linhagem era um tipo normalmente virulento e mortal para a maioria dos animais de laboratorio. As celulas desta linhagem estao envoltas em uma capsula de
polissacarideo
, dando as colonias em aspecto liso, sendo esta linhagem identificada com
S
(
smooth
, em ingles). A outra linhagem de Griffith era um tipo mutante nao virulento que crescia em camundongos. Nesta linhagem, a capa de polissacarideo esta ausente, dando as colonias um aspecto rugoso. Esta linhagem e chamada
R
(
rough
, em ingles).
[
123
]
Griffith inativou algumas celulas virulentas a alta temperatura. Injetou entao as celulas mortas por aquecimento nos camundongos. Os camundongos sobreviveram, mostrando que os restos das celulas nao causam morte. Entretanto os camundongos injetados com uma mistura de celulas virulentas mortas por aquecimento e celulas nao virulentas vivas morreram. Alem disso, as celulas vivas podiam ser recuperadas de camundongos mortos. Estas celulas deram colonias lisas e foram virulentas em uma injecao subsequente. De algum modo, os restos das celulas
S
aquecidas haviam convertido celulas
R
vivas em celulas
S
vivas.
[
124
]
 Streptococcus pneumoniae
Streptococcus pneumoniae
A etapa seguinte era determinar que componente quimico das celulas doadoras mortas havia causado esta conversao. Esta substancia tinha mudado o genotipo da linhagem receptora e portanto podia ser uma candidata a material genetico. Este problema foi resolvido pelos experimentos feitos em 1944 por
Oswald Avery
e dois colegas, C M. Macleod e M. McCarty. Seu enfoque ao problema foi destruir quimicamente todas as principais categorias de substancias no extrato de celulas mortas, uma de cada vez, e descobrir se o extrato havia perdido a habilidade de conversao. As celulas virulentas possuiam uma capa lisa de polissacarideo, enquanto as celulas nao virulentas, nao. Assim os polissacarideos eram um candidato obvio a ser o agente responsavel. Entretanto, quando os polissacarideos foram destruidos, a mistura ainda era capaz de conversao. As proteinas, gorduras e acido ribonucleico (
ARN
) foram todos excluidos. A mistura so perdia a sua capacidade de conversao quando a mistura doadora era tratada com
enzima
desoxirribonuclease (
DNase
), que quebra o ADN. Estes resultados indicavam fortemente que o ADN era o material genetico. Hoje sabemos que os fragmentos do ADN transformante que conferem virulencia entram no cromossomo bacteriano e substituem suas contrapartes que conferem nao virulencia.
[
125
]
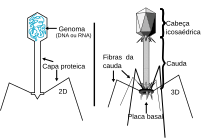 Estrutura do
fago T2
.
Estrutura do
fago T2
.
Os experimentos feitos por
Avery
e seus colegas foram definitivos, mas muitos cientistas mostraram-se muito relutantes em aceitar o ADN (e nao as proteinas) como material genetico.
[
116
]
Evidencias adicionais foram publicadas em 1952 por
Alfred Day Hershey
e
Martha Chase
, cujo experimento com o
fago
T2, um virus que transfecta na bacteria a informacao especifica para a reproducao viral. Se eles pudessem descobrir que material o fago transmitia a bacteria hospedeira, determinariam o material genetico do fago.
[
126
]
O fago tem uma constituicao molecular relativamente simples. A maior parte de sua estrutura e de proteina, com o ADN contido dentro da capa de proteina de sua "cabeca". Hershey e Chase decidiram marcar o ADN e a proteina usando radioisotopos, de modo que pudessem rastrear os dois materiais durante a infeccao. O
fosforo
nao e encontrado nas proteinas mas e uma parte integrante do ADN. Contrariamente, o
enxofre
esta presente nas proteinas mas nunca no ADN. Hershey e Chase incorporaram o radioisotopo de fosforo (
32
P) no ADN do fago e o enxofre (
35
S) nas proteinas de uma cultura separada de fagos. Eles entao infectaram duas culturas de
E. coli
com muitas particulas de virus por celulas: uma cultura de E.
coli
recebeu fagos marcados com
32
P e a outra recebeu fagos marcados com
35
S. Decorrido tempo suficiente para que ocorresse a infeccao, os cientistas removeram as embalagens de fago (chamadas
ghosts
) das celulas bacterianas por agitacao em um
liquidificador
. Eles separaram as celulas bacterianas dos envoltorios dos fagos em uma
centrifuga
e entao mediram a radioatividade nas duas fracoes. Quando o fago marcado com
32
P foi usado para infectar E.
coli
, a mais alta radioatividade foi encontrada dentro das bacterias, indicando que o ADN do fago havia entrado nas celulas. Quando era usado o fago marcado com
35
S, maior parte do material radioativo estava nos involucros dos fagos, indicando que a proteina do fago nunca entrava nas bacterias. A conclusao era inevitavel: o ADN era o material hereditario. As proteinas do fago eram apenas embalagens estruturais abandonadas apos o ADN viral entrar na bacteria.
[
126
]
A investigacao sobre o ADN tem um impacto significativo, especialmente no ambito da
medicina
, mas tambem na agricultura e pecuaria, com objectivos de domesticacao, seleccao e de cruzamentos dirigidos. A moderna biologia e bioquimica fazem uso intensivo da
tecnologia
do
ADN recombinante
, introduzindo genes de interesse em organismos, com o objectivo de expressar uma proteina recombinante concreta, que pode ser:
- isolada para seu uso posterior: por exemplo, podem-se transformar
microorganismos
para produzir grandes quantidades de substancias uteis, como a insulina, que posteriormente se isolam e se utilizam em terapias;
[
127
]
[
128
]
[
129
]
- necessaria para substituir a expressao de um gene endogeno danificado que seja causador de uma patologia, o que permitiria o restabelecimento da actividade da proteina perdida e eventualmente a recuperacao do estado fisiologico normal, nao patologico. Este e o objectivo da
terapia genetica
, um dos campos em que se esta a trabalhar activamente em medicina, analisando vantagens e inconvenientes de diferentes sistemas de administracao do gene (virais e nao virais) e os mecanismos de seleccao do ponto de integracao dos elementos geneticos (distintos para os virus e transposoes) no genoma alvo.
[
130
]
Neste caso, antes de apresentar-se a possibilidade de realizar uma terapia genica numa determinada patologia, e fundamental compreender o impacto do gene de interesse no desenvolvimento de dita patologia, para o qual e necessario o desenvolvimento de um modelo animal, eliminando ou modificando dito gene num animal de laboratorio, mediante a tecnica
nocaute
.
[
131
]
So no caso de os resultados no modelo animal serem satisfatorios podera ser analisada a possibilidade de restabelecer o gene danificado mediante terapia genica;
- utilizada para enriquecer um alimento: por exemplo, a composicao do leite (que e uma importante fonte de proteinas para o consumo humano e animal) pode modificar-se mediante transgenese, adicionando genes exogenos e inactivando genes endogenos para melhorar o seu valor nutricional, reduzir infeccoes nas glandulas mamarias, proporcionar aos consumidores proteinas antipatogenicas e preparar proteinas recombinantes para o uso farmaceutico;
[
132
]
[
133
]
- util para melhorar a resistencia do organismo transformado: por exemplo, em plantas podem-se introduzir genes que conferem resistencia a agentes patogenicos (virus, insectos, fungos), assim como a agentes estressantes abioticos (salinidade, seca, metais pesados).
[
134
]
[
135
]
[
136
]
A
Medicina Forense
pode utilizar o ADN presente no
sangue
, no
semen
, na
pele
, na
saliva
ou em pelos existentes na cena de um crime para identificar o responsavel. Esta tecnica denomina-se
impressao genetica
ou
perfil de ADN
. Ao realizar a impressao genetica, compara-se o comprimento de seccoes altamente variaveis do ADN repetitivo, como os
microssatelites
, entre pessoas diferentes. Este metodo e muito fiavel para identificar um criminoso.
[
137
]
No entanto, a identificacao pode complicar-se se a cena do crime estiver contaminada com ADN de pessoas diferentes.
[
138
]
A tecnica da impressao genetica foi desenvolvida em 1984 pelo geneticista britanico Sir
Alec Jeffreys
,
[
139
]
e utilizada pela primeira vez para condenar
Colin Pitchfork
por causa dos assassinatos de Narborough (Reino Unido) em 1983 e 1986.
[
140
]
Pode-se requerer as pessoas acusadas de certos tipos de crimes que cedam uma amostra de ADN para ser introduzida numa base de dados. Isto tem facilitado o trabalho dos investigadores na resolucao de casos antigos, onde so se obteve uma amostra de ADN da cena do crime, em alguns casos permitindo exonerar um convicto. A impressao genetica tambem pode ser utilizado para identificar vitimas de acidentes em massa,
[
141
]
ou para realizar provas de consanguinidade.
[
142
]
A
Bioinformatica
implica a manipulacao, busca e
extraccao de informacao
dos dados da sequencia do ADN. O desenvolvimento das tecnicas para armazenar e procurar sequencias de ADN gerou avancos no desenvolvimento de
software
para computadores, com muitas aplicacoes, especialmente
algoritmos de busca de frases
,
aprendizagem automatica
e teorias de
bases de dados
.
[
143
]
A busca de frases ou algoritmos de coincidencias, que procuram a ocorrencia de uma sequencia de letras dentro de uma sequencia de letras maior, desenvolveu-se para buscar sequencias especificas de nucleotidos.
[
144
]
Em outras aplicacoes como
editores de textos
, inclusive algoritmos simples podem funcionar, mas as sequencias de ADN podem gerar que estes algoritmos apresentem um comportamento de
quase o pior caso
, devido ao baixo numero de caracteres. O problema relacionado do
alinhamento de sequencias
procura identificar sequencias
homologas
e localizar
mutacoes
especificas que as diferenciam. Estas tecnicas, fundamentalmente o
alinhamento multiplo de sequencias
, utilizam-se ao estudar as relacoes
filogeneticas
e a funcao das proteinas.
[
145
]
As coleccoes de dados que representam sequencias do ADN do tamanho de um genoma, tais como as produzidas pelo
Projecto Genoma Humano
, sao dificeis de utilizar sem notacoes que marcam a localizacao dos genes e dos elementos reguladores em cada cromossoma. As regioes de ADN que tem padroes associados com genes codificantes de proteinas ou ARN podem identificar-se por algoritmos de
localizacao de genes
, o que permite aos investigadores predizer a presenca de
produtos genicos
especificos num organismo mesmo antes que se tenha isolado experimentalmente.
[
146
]
A nanotecnologia de ADN utiliza as propriedades unicas de reconhecimento molecular de ADN e outros acidos nucleicos para criar complexos ramificados auto-ensamblados com propriedades uteis. Neste caso, o ADN utiliza-se como um material estrutural, mais que como um portador de informacao biologica.
[
147
]
Isto conduziu a criacao de laminas periodicas de duas dimensoes (ambas baseadas em azulejos, assim como usando o metodo de "ADN origami"), para alem de estruturas em tres dimensoes em forma de
poliedros
.
[
148
]
O ADN armazena mutacoes conservadas com o tempo e portanto contem informacao historica. Comparando sequencias de ADN, os geneticistas podem inferir a historia evolutiva dos organismos, a sua
filogenia
.
[
149
]
O campo da filogenia e uma ferramenta potente na
biologia evolutiva
. Se se compararem as sequencias de ADN dentro de uma especie, os
geneticistas de populacoes
podem conhecer a historia de populacoes particulares. Isto pode-se utilizar numa ampla variedade de estudos, desde
ecologia
ate
antropologia
; por exemplo, evidencia baseada na analise de ADN esta a ser utilizada para identificar as Dez Tribos Perdidas de Israel.
[
150
]
[
151
]
Notas
- ↑
Apesar do uso da sigla em ingles ser bem comum no Brasil, legisladores brasileiros preferem o uso da sigla em portugues em leis brasileiras
[
1
]
Referencias
- ↑
https://www.planalto.gov.br/ccivil_03/_ato2004-2006/2005/lei/l11105.htm
?
- ↑
≪DNA is held together by hydrophobic forces≫
.
Tech Explorist
(em ingles). 23 de setembro de 2019
. Consultado em 23 de setembro de 2019
.
Copia arquivada em 23 de setembro de 2019
- ↑
a
b
c
d
e
f
Bruce Alberts,Alexander Johnson, Julian Lewis, Kazuo, Martin Raff, Keith Roberts, and Peter Walters (2002).
Molecular Biology of the Cell; Fourth Edition
. Nova Iorque e Londres: Garland Science.
ISBN
0-8153-3218-1
. Consultado em 25 de junho de 2007
.
Copia arquivada em 3 de outubro de 2009
- ↑
Butler, John M. (2001)
Forensic DNA Typing
"Elsevier". pp. 14?15.
ISBN 978-0-12-147951-0
.
- ↑
Mandelkern M, Elias J, Eden D, Crothers D (1981). ≪The dimensions of DNA in solution≫.
J Mol Biol
.
152
(1): 153?61.
PMID
7338906
- ↑
Gregory S; et al. (2006). ≪The DNA sequence and biological annotation of human chromosome 1≫.
Nature
.
441
(7091): 315?21.
PMID
16710414
- ↑
Watson J, Crick F (1953).
≪Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid≫
(PDF)
.
Nature
.
171
(4356): 737?8.
PMID
13054692
. Consultado em 25 de junho de 2007
.
Copia arquivada
(PDF)
em 23 de agosto de 2014
- ↑
Berg J., Tymoczko J. and Stryer L. (2002)
Biochemistry.
W. H. Freeman and Company
ISBN 0-7167-4955-6
- ↑
≪Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB≫
(em ingles). Commission on Biochemical Nomenclature (CBN)
. Consultado em 3 de janeiro de 2006
.
Copia arquivada em 5 de fevereiro de 2007
- ↑
G. Vieira, H. de Lencastre , L. Archer (1989). ≪Restriction analysis of PBS 1-related phages≫. Springer-Verlag.
Archives of Virology
.
106
.
doi
:
10.1007/BF01311043
- ↑
Ponnuswamy P, Gromiha M (1994). ≪On the conformational stability of oligonucleotide duplexes and tRNA molecules≫.
J Theor Biol
.
169
(4): 419?32.
PMID
7526075
- ↑
Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000).
≪Mechanical stability of single DNA molecules≫
.
Biophys J
.
78
(4): 1997?2007.
PMID
10733978
. Consultado em 10 de novembro de 2007
.
Copia arquivada
(PDF)
em 24 de setembro de 2019
- ↑
Chalikian T, Volker J, Plum G, Breslauer K (1999).
≪A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques≫
.
Proc Natl Acad Sci U S A
.
96
(14): 7853?8.
PMID
10393911
. Consultado em 10 de novembro de 2007
.
Copia arquivada
(PDF)
em 24 de setembro de 2019
- ↑
Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). ≪Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern≫.
Biochemistry
.
43
(51): 15996?6010.
PMID
15609994
- ↑
G. Michael Blackburn, Michael J. Gait, David Loakes, David M. Williams (2006).
Nucleic acids in chemistry and biology
3 ed. [S.l.]: Royal Society of Chemistry.
ISBN
978-0-85404-654-6
- ↑
Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). ≪Crystal structure analysis of a complete turn of B-DNA≫.
Nature
.
287
(5784): 755?8.
PMID
7432492
- ↑
Pabo C, Sauer R (1984). ≪Protein-DNA recognition≫.
Annu Rev Biochem
.
53
: 293?321.
PMID
6236744
- ↑
Makalowska I, Lin C, Makalowski W (2005). ≪Overlapping genes in vertebrate genomes≫.
Comput Biol Chem
.
29
(1): 1?12.
PMID
15680581
- ↑
Johnson Z, Chisholm S (2004). ≪Properties of overlapping genes are conserved across microbial genomes≫.
Genome Res
.
14
(11): 2268?72.
PMID
15520290
- ↑
Lamb R, Horvath C (1991). ≪Diversity of coding strategies in influenza viruses≫.
Trends Genet
.
7
(8): 261?6.
PMID
1771674
- ↑
Benham C, Mielke S (2005). ≪DNA mechanics≫.
Annu Rev Biomed Eng
.
7
: 21?53.
PMID
16004565
- ↑
a
b
Champoux J (2001). ≪DNA topoisomerases: structure, function, and mechanism≫.
Annu Rev Biochem
.
70
: 369?413.
PMID
11395412
- ↑
a
b
Wang J (2002). ≪Cellular roles of DNA topoisomerases: a molecular perspective≫.
Nat Rev Mol Cell Biol
.
3
(6): 430?40.
PMID
12042765
- ↑
a
b
Hayashi G, Hagihara M, Nakatani K (2005). ≪Application of L-DNA as a molecular tag≫.
Nucleic Acids Symp Ser (Oxf)
.
49
: 261?262.
PMID
17150733
- ↑
Vargason JM, Eichman BF, Ho PS (2000). ≪The extended and eccentric E-DNA structure induced by cytosine methylation or bromination≫.
Nature Structural Biology
.
7
: 758?761.
PMID
10966645
- ↑
Wang G, Vasquez KM (2006). ≪Non-B DNA structure-induced genetic instability≫.
Mutat Res
.
598
(1?2): 103?119.
PMID
16516932
- ↑
Allemand; et al. (1998). ≪Stretched and overwound DNA forms a Pauling-like structure with exposed bases≫.
PNAS
.
24
: 14152-14157.
PMID
9826669
- ↑
Palecek E (1991). ≪Local supercoil-stabilized DNA structures≫.
Critical Reviews in Biochemistry and Molecular Biology
.
26
(2): 151?226.
PMID
1914495
- ↑
Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). ≪Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies≫.
J Biomol Struct Dyn
.
6
(2): 299?309.
PMID
2482766
- ↑
Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). ≪Polymorphism of DNA double helices≫.
J. Mol. Biol
.
143
(1): 49?72.
PMID
7441761
- ↑
Wahl M, Sundaralingam M (1997). ≪Crystal structures of A-DNA duplexes≫.
Biopolymers
.
44
(1): 45?63.
PMID
9097733
- ↑
Lu XJ, Shakked Z, Olson WK (2000). ≪A-form conformational motifs in ligand-bound DNA structures≫.
J. Mol. Biol
.
300
(4): 819-40.
PMID
10891271
- ↑
Rothenburg S, Koch-Nolte F, Haag F. ≪DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles≫.
Immunol Rev
.
184
: 286?98.
PMID
12086319
- ↑
Oh D, Kim Y, Rich A (2002).
≪Z-DNA-binding proteins can act as potent effectors of gene expression in vivo≫
.
Proc. Natl. Acad. Sci. U.S.A
.
99
(26): 16666-71.
PMID
12486233
- ↑
≪NDB UD0017≫
. Ndbserver.rutgers.edu
. Consultado em 25 de junho de 2008
. Arquivado do
original
em 7 de junho de 2013
- ↑
a
b
Greider C, Blackburn E (1985). ≪Identification of a specific telomere terminal transferase activity in Tetrahymena extracts≫.
Cell
.
43
(2 Pt 1): 405?13.
PMID
3907856
- ↑
a
b
c
Nugent C, Lundblad V (1998).
≪The telomerase reverse transcriptase: components and regulation≫
.
Genes Dev
.
12
(8): 1073?85.
PMID
9553037
. Consultado em 24 de junho de 2008
.
Copia arquivada em 27 de setembro de 2007
- ↑
Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997).
≪Normal human chromosomes have long G-rich telomeric overhangs at one end≫
.
Genes Dev
.
11
(21): 2801?9.
PMID
9353250
. Consultado em 24 de junho de 2008
.
Copia arquivada em 27 de setembro de 2007
- ↑
a
b
Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006).
≪Quadruplex DNA: sequence, topology and structure≫
.
Nucleic Acids Res
.
34
(19): 5402?15.
PMID
17012276
.
doi
:
10.1093/nar/gkl655
. Consultado em 24 de junho de 2008
.
Copia arquivada em 24 de setembro de 2019
- ↑
Parkinson G, Lee M, Neidle S (2002). ≪Crystal structure of parallel quadruplexes from human telomeric DNA≫.
Nature
.
417
(6891): 876?80.
PMID
12050675
.
doi
:
10.1038/nature755
- ↑
Huppert, Julian Leon (15 de dezembro de 2007).
≪Four-stranded DNA: cancer, gene regulation and drug development≫
.
Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences
.
365
(1861): 2969 -2984.
doi
:
10.1098/rsta.2007.0011
. Consultado em 8 de outubro de 2010
.
Copia arquivada em 8 de dezembro de 2015
- ↑
Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). ≪Mammalian telomeres end in a large duplex loop≫.
Cell
.
97
(4): 503?14.
PMID
10338214
- ↑
Klose R, Bird A (2006). ≪Genomic DNA methylation: the mark and its mediators≫.
Trends Biochem Sci
.
31
(2): 89?97.
PMID
16403636
.
doi
:
10.1016/j.tibs.2005.12.008
- ↑
Bird A (2002). ≪DNA methylation patterns and epigenetic memory≫.
Genes Dev
.
16
(1): 6?21.
PMID
11782440
.
doi
:
10.1101/gad.947102
- ↑
Walsh C, Xu G (2006). ≪Cytosine methylation and DNA repair≫.
Curr Top Microbiol Immunol
.
301
: 283?315.
PMID
16570853
.
doi
:
10.1007/3-540-31390-7_11
- ↑
Ratel D, Ravanat J, Berger F, Wion D (2006). ≪N6-methyladenine: the other methylated base of DNA≫.
Bioessays
.
28
(3): 309?15.
PMID
16479578
.
doi
:
10.1002/bies.20342
- ↑
Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). ≪beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei≫.
Cell
.
75
(6): 1129?36.
PMID
8261512
.
doi
:
10.1016/0092-8674(93)90322-H
- ↑
≪PDB 1JDG≫
. Rcsb.org
. Consultado em 16 de julho de 2008
.
Copia arquivada em 22 de setembro de 2008
- ↑
Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). ≪Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation≫.
Biochemistry
.
42
(30): 9221?6.
PMID
12885257
.
doi
:
10.1021/bi034593c
- ↑
Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). ≪Hydroxyl radicals and DNA base damage≫.
Mutat Res
.
424
(1?2): 9?21.
PMID
10064846
- ↑
Shigenaga M, Gimeno C, Ames B (1989).
≪Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of
in vivo
oxidative DNA damage≫
.
Proc Natl Acad Sci U S A
.
86
(24): 9697?701.
PMID
2602371
.
doi
:
10.1073/pnas.86.24.9697
. Consultado em 16 de julho de 2008
.
Copia arquivada em 7 de marco de 2008
- ↑
Cathcart R, Schwiers E, Saul R, Ames B (1984).
≪Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage≫
(PDF)
.
Proc Natl Acad Sci U S A
.
81
(18): 5633?7.
PMID
6592579
.
doi
:
10.1073/pnas.81.18.5633
. Consultado em 16 de julho de 2008
.
Copia arquivada
(PDF)
em 25 de junho de 2008
- ↑
Valerie K, Povirk L (2003). ≪Regulation and mechanisms of mammalian double-strand break repair≫.
Oncogene
.
22
(37): 5792?812.
PMID
12947387
.
doi
:
10.1038/sj.onc.1206679
- ↑
Ferguson L, Denny W (1991). ≪The genetic toxicology of acridines≫.
Mutat Res
.
258
(2): 123?60.
PMID
1881402
- ↑
Jeffrey A (1985). ≪DNA modification by chemical carcinogens≫.
Pharmacol Ther
.
28
(2): 237?72.
PMID
3936066
.
doi
:
10.1016/0163-7258(85)90013-0
- ↑
Stephens T, Bunde C, Fillmore B (2000). ≪Mechanism of action in thalidomide teratogenesis≫.
Biochem Pharmacol
.
59
(12): 1489?99.
PMID
10799645
.
doi
:
10.1016/S0006-2952(99)00388-3
- ↑
Brana M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). ≪Intercalators as anticancer drugs≫.
Curr Pharm Des
.
7
(17): 1745?80.
PMID
11562309
.
doi
:
10.2174/1381612013397113
- ↑
Venter J; et al. (2001). ≪The sequence of the human genome≫.
Science
.
291
(5507): 1304?51.
PMID
11181995
.
doi
:
10.1126/science.1058040
- ↑
Criada a partir de.
≪PDB 1MSW≫
. Rcsb.org
. Consultado em 4 de setembro de 2008
.
Copia arquivada em 6 de janeiro de 2008
- ↑
Thanbichler M, Wang S, Shapiro L (2005). ≪The bacterial nucleoid: a highly organized and dynamic structure≫.
J Cell Biochem
.
96
(3): 506?21.
PMID
15988757
.
doi
:
10.1002/jcb.20519
- ↑
Wolfsberg T, McEntyre J, Schuler G (2001). ≪Guide to the draft human genome≫.
Nature
.
409
(6822): 824?6.
PMID
11236998
.
doi
:
10.1038/35057000
- ↑
Gregory T (2005).
≪The C-value enigma in plants and animals: a review of parallels and an appeal for partnership≫
.
Ann Bot (Lond)
.
95
(1): 133?46.
PMID
15596463
.
doi
:
10.1093/aob/mci009
. Consultado em 10 de novembro de 2007
.
Copia arquivada em 16 de maio de 2007
- ↑
The ENCODE Project Consortium (2007). ≪Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project≫.
Nature
.
447
(7146): 799?816.
doi
:
10.1038/nature05874
- ↑
Pidoux A, Allshire R (2005).
≪The role of heterochromatin in centromere function≫
.
Philos Trans R Soc Lond B Biol Sci
.
360
(1455): 569?79.
PMID
15905142
.
doi
:
10.1098/rstb.2004.1611
- ↑
Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002).
≪Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22≫
.
Genome Res
.
12
(2): 272?80.
PMID
11827946
.
doi
:
10.1101/gr.207102
. Consultado em 10 de novembro de 2007
.
Copia arquivada em 28 de outubro de 2007
- ↑
Harrison P, Gerstein M (2002). ≪Studying genomes through the aeons: protein families, pseudogenes and proteome evolution≫.
J Mol Biol
.
318
(5): 1155?74.
PMID
12083509
.
doi
:
10.1016/S0022-2836(02)00109-2
- ↑
≪Genetic Code≫
. Brooklyn.cuny.edu
. Consultado em 7 de outubro de 2010
.
Copia arquivada em 9 de novembro de 2010
- ↑
≪Transcribe and Translate a Gene≫
. Learn.genetics.utah.edu
. Consultado em 7 de outubro de 2010
. Arquivado do
original
em 1 de setembro de 2010
- ↑
Alba M (2001).
≪Replicative DNA polymerases≫
.
Genome Biol
.
2
(1): REVIEWS3002.
PMID
11178285
.
doi
:
10.1186/gb-2001-2-1-reviews3002
- ↑
Sandman K, Pereira S, Reeve J (1998). ≪Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome≫.
Cell Mol Life Sci
.
54
(12): 1350?64.
PMID
9893710
.
doi
:
10.1007/s000180050259
- ↑
Dame RT (2005). ≪The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin≫.
Mol. Microbiol
.
56
(4): 858?70.
PMID
15853876
.
doi
:
10.1111/j.1365-2958.2005.04598.x
- ↑
Luger K, Mader A, Richmond R, Sargent D, Richmond T (1997). ≪Crystal structure of the nucleosome core particle at 2.8 A resolution≫.
Nature
.
389
(6648): 251?60.
PMID
9305837
.
doi
:
10.1038/38444
- ↑
Jenuwein T, Allis C (2001). ≪Translating the histone code≫.
Science
.
293
(5532): 1074?80.
PMID
11498575
.
doi
:
10.1126/science.1063127
- ↑
Ito T. ≪Nucleosome assembly and remodelling≫.
Curr Top Microbiol Immunol
.
274
: 1?22.
PMID
12596902
- ↑
Thomas J (2001). ≪HMG1 and 2: architectural DNA-binding proteins≫.
Biochem Soc Trans
.
29
(Pt 4): 395?401.
PMID
11497996
.
doi
:
10.1042/BST0290395
- ↑
Grosschedl R, Giese K, Pagel J (1994). ≪HMG domain proteins: architectural elements in the assembly of nucleoprotein structures≫.
Trends Genet
.
10
(3): 94?100.
PMID
8178371
.
doi
:
10.1016/0168-9525(94)90232-1
- ↑
Iftode C, Daniely Y, Borowiec J (1999). ≪Replication protein A (RPA): the eukaryotic SSB≫.
Crit Rev Biochem Mol Biol
.
34
(3): 141?80.
PMID
10473346
.
doi
:
10.1080/10409239991209255
- ↑
Criado a partir de.
≪PDB 1LMB≫
. Rcsb.org
. Consultado em 30 de setembro de 2008
.
Copia arquivada em 6 de janeiro de 2008
- ↑
Myers L, Kornberg R (2000). ≪Mediator of transcriptional regulation≫.
Annu Rev Biochem
.
69
: 729?49.
PMID
10966474
.
doi
:
10.1146/annurev.biochem.69.1.729
- ↑
Spiegelman B, Heinrich R (2004). ≪Biological control through regulated transcriptional coactivators≫.
Cell
.
119
(2): 157?67.
PMID
15479634
.
doi
:
10.1016/j.cell.2004.09.037
- ↑
Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003).
≪A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells≫
.
Proc Natl Acad Sci USA
.
100
(14): 8164?9.
PMID
12808131
.
doi
:
10.1073/pnas.1332764100
. Consultado em 30 de setembro de 2008
.
Copia arquivada em 24 de setembro de 2019
- ↑
Pabo C, Sauer R (1984). ≪Protein-DNA recognition≫.
Annu Rev Biochem
.
53
: 293?321.
PMID
6236744
.
doi
:
10.1146/annurev.bi.53.070184.001453
- ↑
Criado a partir de.
≪PDB 1RVA≫
. Rcsb.org
. Consultado em 30 de setembro de 2008
.
Copia arquivada em 6 de janeiro de 2008
- ↑
Bickle T, Kruger D (1993).
≪Biology of DNA restriction≫
.
Microbiol Rev
.
57
(2): 434?50.
PMID
8336674
- ↑
a
b
Doherty A, Suh S (2000).
≪Structural and mechanistic conservation in DNA ligases.≫
.
Nucleic Acids Res
.
28
(21): 4051?8.
PMID
11058099
.
doi
:
10.1093/nar/28.21.4051
. Consultado em 30 de setembro de 2008
.
Copia arquivada em 24 de setembro de 2019
- ↑
Schoeffler A, Berger J (2005). ≪Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism≫.
Biochem Soc Trans
.
33
(Pt 6): 1465?70.
PMID
16246147
.
doi
:
10.1042/BST20051465
- ↑
Tuteja N, Tuteja R (2004). ≪Unraveling DNA helicases. Motif, structure, mechanism and function≫.
Eur J Biochem
.
271
(10): 1849?63.
PMID
15128295
.
doi
:
10.1111/j.1432-1033.2004.04094.x
- ↑
Joyce C, Steitz T (1995).
≪Polymerase structures and function: variations on a theme?≫
.
J Bacteriol
.
177
(22): 6321?9.
PMID
7592405
. Consultado em 30 de setembro de 2008
.
Copia arquivada
(PDF)
em 24 de setembro de 2019
- ↑
Hubscher U, Maga G, Spadari S (2002). ≪Eukaryotic DNA polymerases≫.
Annu Rev Biochem
.
71
: 133?63.
PMID
12045093
.
doi
:
10.1146/annurev.biochem.71.090501.150041
- ↑
Johnson A, O'Donnell M (2005). ≪Cellular DNA replicases: components and dynamics at the replication fork≫.
Annu Rev Biochem
.
74
: 283?315.
PMID
15952889
.
doi
:
10.1146/annurev.biochem.73.011303.073859
- ↑
Tarrago-Litvak L, Andreola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994).
≪The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention≫
.
FASEB J
.
8
(8): 497?503.
PMID
7514143
. Consultado em 30 de setembro de 2008
.
Copia arquivada em 5 de setembro de 2008
- ↑
Martinez E (2002). ≪Multi-protein complexes in eukaryotic gene transcription≫.
Plant Mol Biol
.
50
(6): 925?47.
PMID
12516863
.
doi
:
10.1023/A:1021258713850
- ↑
Criado a partir de.
≪PDB 1M6G≫
. Rcsb.org
. Consultado em 17 de outubro de 2008
.
Copia arquivada em 10 de janeiro de 2010
- ↑
Cremer T, Cremer C (2001). ≪Chromosome territories, nuclear architecture and gene regulation in mammalian cells≫.
Nat Rev Genet
.
2
(4): 292?301.
PMID
11283701
.
doi
:
10.1038/35066075
- ↑
≪Chromosomal crossover≫
(em ingles). Sciencedaily.com
. Consultado em 7 de outubro de 2010
. Arquivado do
original
em 21 de maio de 2011
- ↑
Pal C, Papp B, Lercher M (2006). ≪An integrated view of protein evolution≫.
Nat Rev Genet
.
7
(5): 337?48.
PMID
16619049
.
doi
:
10.1038/nrg1838
- ↑
O'Driscoll M, Jeggo P (2006). ≪The role of double-strand break repair - insights from human genetics≫.
Nat Rev Genet
.
7
(1): 45?54.
PMID
16369571
.
doi
:
10.1038/nrg1746
- ↑
Vispe S, Defais M (1997). ≪Mammalian Rad51 protein: a RecA homologue with pleiotropic functions≫.
Biochimie
.
79
(9-10): 587?92.
PMID
9466696
.
doi
:
10.1016/S0300-9084(97)82007-X
- ↑
Neale MJ, Keeney S (2006). ≪Clarifying the mechanics of DNA strand exchange in meiotic recombination≫.
Nature
.
442
(7099): 153?8.
PMID
16838012
.
doi
:
10.1038/nature04885
- ↑
Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). ≪The RuvABC resolvasome≫.
Eur J Biochem
.
269
(22): 5492?501.
PMID
12423347
.
doi
:
10.1046/j.1432-1033.2002.03250.x
- ↑
Joyce G (2002). ≪The antiquity of RNA-based evolution≫.
Nature
.
418
(6894): 214?21.
PMID
12110897
.
doi
:
10.1038/418214a
- ↑
Orgel L.
≪Prebiotic chemistry and the origin of the RNA world≫
(PDF)
.
Crit Rev Biochem Mol Biol
.
39
(2): 99?123.
PMID
15217990
.
doi
:
10.1080/10409230490460765
. Consultado em 10 de agosto de 2010
.
Copia arquivada
(PDF)
em 13 de novembro de 2018
- ↑
Davenport R (2001). ≪Ribozymes. Making copies in the RNA world≫.
Science
.
292
(5520). 1278 paginas.
PMID
11360970
.
doi
:
10.1126/science.292.5520.1278a
- ↑
Szathmary E (1992).
≪What is the optimum size for the genetic alphabet?≫
(PDF)
.
Proc Natl Acad Sci U S A
.
89
(7): 2614?8.
PMID
1372984
.
doi
:
10.1073/pnas.89.7.2614
. Consultado em 17 de outubro de 2008
.
Copia arquivada
(PDF)
em 25 de junho de 2008
- ↑
Lindahl T (1993). ≪Instability and decay of the primary structure of DNA≫.
Nature
.
362
(6422): 709?15.
PMID
8469282
.
doi
:
10.1038/362709a0
- ↑
Vreeland R, Rosenzweig W, Powers D (2000). ≪Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal≫.
Nature
.
407
(6806): 897?900.
PMID
11057666
.
doi
:
10.1038/35038060
- ↑
Hebsgaard M, Phillips M, Willerslev E (2005). ≪Geologically ancient DNA: fact or artefact?≫.
Trends Microbiol
.
13
(5): 212?20.
PMID
15866038
.
doi
:
10.1016/j.tim.2005.03.010
- ↑
Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002). ≪Curiously modern DNA for a "250 million-year-old" bacterium≫.
J Mol Evol
.
54
(1): 134?7.
PMID
11734907
.
doi
:
10.1007/s00239-001-0025-x
- ↑
Birnbaum D, Coulier F, Pebusque MJ, Pontarotti P. (2000). ≪
"
Paleogenomics": looking in the past to the future≫.
J Exp Zool
.
288
((1): 21-2).
PMID
10750049
- ↑
Blanchette M, Green ED, Miller W, Haussler D. (2004). ≪Reconstructing large regions of an ancestral mammalian genome in silico≫.
Genome Res
.
14
((12): 2412-23).
PMID
15574820
.
doi
:
10.1101/gr.2800104
- ↑
Gaucher EA, Thomson JM, Burgan MF, Benner SA. (2003). ≪Inferring the palaeoenvironment of ancient bacteria on the basis of resurrected proteins≫.
Nature
.
425
((6955): 285-8).
PMID
13679914
.
doi
:
10.1038/nature01977
- ↑
Thornton JW. (2004). ≪Resurrecting ancient genes: experimental analysis of extinct molecules≫.
Nat Rev Genet
.
5
((5): 366-75).
PMID
15143319
.
doi
:
10.1038/nrg1324
- ↑
Benner SA, Caraco MD, Thomson JM, Gaucher EA. (2002). ≪Planetary biology--paleontological, geological, and molecular histories of life≫.
Science
.
296
((5569): 864-8).
PMID
11988562
.
doi
:
10.1126/science.1069863
- ↑
a
b
c
d
Dahm R (Janeiro de 2008).
≪Discovering DNA: Friedrich Miescher and the early years of nucleic acid research≫
(PDF)
.
Human Genetics
.
122
(6): 565?581.
PMID
17901982
.
doi
:
10.1007/s00439-007-0433-0
. Consultado em 25 de agosto de 2010
. Arquivado do
original
(PDF)
em 19 de janeiro de 2012
- ↑
Morange, Michel. A History of Molecular Biology. Cambridge, MA: Harvard University Press. 1998.
- ↑
a
b
Gerald Karp (2005).
Biologia celular e molecular
. conceitos e experimentos. [S.l.]: Editora Manole. 9788520415931
- ↑
Mary Ellen Jones (29 de setembro de 1953).
≪Albrecht Kossel, A Biographical Sketch≫
(PDF)
.
The Yale Journal of Biology and Medicine
.
26
.
PMID
13103145
. Consultado em 23 de agosto de 2010
.
Copia arquivada
(PDF)
em 9 de fevereiro de 2019
- ↑
Graeme K. Hunter (2000).
Vital forces
. the discovery of the molecular basis of life. [S.l.]: Academic Press. 9780123618115
- ↑
≪The Nobel Prize in Physiology or Medicine 1910≫
.
Site oficial da Fundacao Nobel
(em ingles). Nobelprize.org
. Consultado em 23 de agosto de 2010
.
Copia arquivada em 12 de outubro de 2010
- ↑
Levene P (1 de dezembro de 1919).
≪The structure of yeast nucleic acid≫
.
J Biol Chem
.
40
(2): 415?24
. Consultado em 9 de outubro de 2010
.
Copia arquivada em 29 de junho de 2009
- ↑
Levene, P; London, E.S. (6 de fevereiro de 1929).
≪GUANINEDESOXYPENTOSIDE FROM THYMUS NUCLEIC ACID≫
(PDF)
.
J Biol Chem
.
81
(3): 711?13
. Consultado em 9 de outubro de 2010
.
Copia arquivada
(PDF)
em 11 de junho de 2019
- ↑
Levene, P; London, E.S. (17 de junho de 1929).
≪THE STRUCTURE OF THYMONUCLEIC ACID≫
(PDF)
.
J Biol Chem
.
83
(3): 793-802
. Consultado em 9 de outubro de 2010
.
Copia arquivada
(PDF)
em 11 de junho de 2019
- ↑
Clare O'Connor (2008).
≪Isolating Hereditary Material: Frederick Griffith, Oswald Avery, Alfred Hershey, and Martha Chase≫
(em ingles).
Nature
. Consultado em 25 de agosto de 2010
.
Copia arquivada em 8 de agosto de 2010
- ↑
Lehrer, Steven (2006).
Explorers of the Body
2ª ed. [S.l.: s.n.] pp. 47?52
. Consultado em 20 de maio de 2009
.
Copia arquivada em 14 de novembro de 2013
- ↑
Avery, O T; Macleod C M, McCarty M (outubro 2000). "Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from Pneumococcus type III. Oswald Theodore Avery (1877-1955)". Clin. Orthop. Relat. Res. 379 (379 Suppl): S3?8. doi:10.1097/00003086-200010001-00002.
PMID 11039746
.
- ↑
a
b
A. D. Hershey and Martha Chase (20 de setembro de 1952).
≪INDEPENDENT FUNCTIONS OF VIRAL PROTEIN AND NUCLEIC ACID IN GROWTH OF BACTERIOPHAGE≫
(PDF)
. Rockefeller University Press.
The Journal of Cell Biology
(em ingles).
36
(1): 39-56
. Consultado em 12 de agosto de 2010
.
Copia arquivada
(PDF)
em 15 de julho de 2010
- ↑
Miller WL. (1979).
≪Use of recombinant DNA technology for the production of polypeptides≫
.
Adv Exp Med Biol
.
118
: 153-74
. Consultado em 17 de outubro de 2008
.
Copia arquivada em 13 de fevereiro de 2009
- ↑
Leader B, Baca QJ, Golan DE. (2008).
≪Protein therapeutics: a summary and pharmacological classification≫
.
Nat Rev Drug Discov
.
7
(1): 21-39
. Consultado em 17 de outubro de 2008
.
Copia arquivada em 6 de janeiro de 2009
- ↑
Dingermann T. (2008).
≪Recombinant therapeutic proteins: production platforms and challenges≫
.
Biotechnol J
.
3
(1): 90-7
[ligacao inativa]
- ↑
Voigt K, Izsvak Z, Ivics Z. (2008).
≪Targeted gene insertion for molecular medicine≫
.
J Mol Med
. Jul 8
. Consultado em 17 de outubro de 2008
. Arquivado do
original
em 24 de setembro de 2019
- ↑
Houdebine L (2007). ≪Transgenic animal models in biomedical research≫.
Methods Mol Biol
.
360
: 163?202.
PMID
17172731
[https://web.archive.org/web/20190114135326/http://www.ncbi.nlm.nih.gov/pubmed/17172731?ordinalpos=6&itool=EntrezSystem2.PEntrez.Pubmed.Pubmed_ResultsPanel.Pubmed_RVDocSum%7D%7D Arquivado em
14 de janeiro de 2019, no
Wayback Machine
.
- ↑
Soler E, Thepot D, Rival-Gervier S, Jolivet G, Houdebine LM. (2006). ≪Preparation of recombinant proteins in milk to improve human and animal health≫.
Reprod Nutr Dev
.
46
((5) 579-88)
[1]
Arquivado em
3 de julho de 2009, no
Wayback Machine
.
- ↑
Chavez A, Munoz de Chavez M. (2003). ≪Nutrigenomics in public health nutrition: short-term perspectives≫.
Eur J Clin Nutr
.
57
(Suppl 1 S97-100)
[2]
Arquivado em
6 de janeiro de 2009, no
Wayback Machine
.
- ↑
Vasil IK. (2007).
≪Molecular genetic improvement of cereals: transgenic wheat (Triticum aestivum L.)≫
.
Plant Cell Rep
.
26
(8): 1133-54
. Consultado em 17 de outubro de 2008
. Arquivado do
original
em 24 de setembro de 2019
- ↑
Daniell H, Dhingra A (2002). ≪Multigene engineering: dawn of an exciting new era in biotechnology≫.
Curr Opin Biotechnol
.
13
(2): 136?41.
PMID
11950565
.
doi
:
10.1016/S0958-1669(02)00297-5
- ↑
Job D (2002). ≪Plant biotechnology in agriculture≫.
Biochimie
.
84
(11): 1105?10.
PMID
12595138
.
doi
:
10.1016/S0300-9084(02)00013-5
- ↑
Collins A, Morton N (1994).
≪Likelihood ratios for DNA identification≫
(PDF)
.
Proc Natl Acad Sci USA
.
91
(13): 6007?11.
PMID
8016106
.
doi
:
10.1073/pnas.91.13.6007
. Consultado em 18 de outubro de 2008
.
Copia arquivada
(PDF)
em 27 de novembro de 2007
- ↑
Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J (1997). ≪Interpreting DNA mixtures≫.
J Forensic Sci
.
42
(2): 213?22.
PMID
9068179
- ↑
Jeffreys A, Wilson V, Thein S (1985). ≪Individual-specific 'fingerprints' of human DNA≫.
Nature
.
316
(6023): 76?9.
PMID
2989708
.
doi
:
10.1038/316076a0
- ↑
≪Colin Pitchfork ? first murder conviction on DNA evidence also clears the prime suspect Forensic Science Service≫
(em ingles). Forensic.gov.uk
. Consultado em 23 de dezembro de 2006
. Arquivado do
original
em 10 de janeiro de 2012
- ↑
≪DNA Identification in Mass Fatality Incidents≫
.
National Institute of Justice
. Massfatality.dna.gov. Setembro de 2006. Arquivado do
original
em 12 de novembro de 2006
- ↑
Bhattacharya, Shaoni. (20 de Abril de 2004).
≪Killer convicted thanks to relative's DNA
≫
. Newscientist.com
. Consultado em 22 de dezembro de 2006
.
Copia arquivada em 12 de fevereiro de 2007
- ↑
Baldi, Pierre. Brunak, Soren (2001).
Bioinformatics: The Machine Learning Approach
. [S.l.]: MIT Press.
ISBN
978-0-262-02506-5
- ↑
Gusfield, Dan.
Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology
. Cambridge University Press, 15 de janeiro de 1997.
ISBN 978-0-521-58519-4
.
- ↑
Sjolander K (2004).
≪Phylogenomic inference of protein molecular function: advances and challenges≫
.
Bioinformatics
.
20
(2): 170?9.
PMID
14734307
.
doi
:
10.1093/bioinformatics/bth021
. Consultado em 18 de outubro de 2008
.
Copia arquivada em 11 de outubro de 2008
- ↑
Mount, DM (2004).
Bioinformatics: Sequence and Genome Analysis
2 ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
ISBN
0879697121
- ↑
Yin P, Hariadi RF, Sahu S, Choi HMT, Park SH, LaBean T, Reif JH. (2008).
≪Programming DNA Tube Circumferences≫
.
Science
.
321
(5890): 824 - 826
- ↑
Andersen ES, Dong M, Nielsen MM (maio de 2009). ≪Self-assembly of a nanoscale DNA box with a controllable lid≫.
Nature
.
459
(7243): 73?6.
PMID
19424153
.
doi
:
10.1038/nature07971
- ↑
Wray G (2002).
≪Dating branches on the tree of life using DNA≫
.
Genome Biol
.
3
(1): REVIEWS0001.
PMID
11806830
.
doi
:
10.1046/j.1525-142X.1999.99010.x
. Consultado em 18 de outubro de 2008
.
Copia arquivada em 24 de setembro de 2019
- ↑
Lost Tribes of Israel
,
NOVA
, data de transmissao na PBS: 22 de Fevereiro de 2000. Transcricao disponivel em
PBS.org,
Arquivado em
16 de setembro de 2008, no
Wayback Machine
.. Pagina acedida a 4 de Marco de 2006.
- ↑
Kleiman, Yaakov. (13 de Janeiro de 2000).
≪The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition≫
. Aish.com
. Consultado em 4 de Marco de 2006
.
Copia arquivada em 25 de abril de 2016
- Introducao a genetica, Riffiths, Wessler, Lewontin, Gesbart, suzuki, Miller, 8º Edicao,
Guanabara Koogan
, 2006.
- Biologia; Jose Mariano Amabis, Gilberto Rodriges Martho; Moderna; 2004
 Commons
Commons