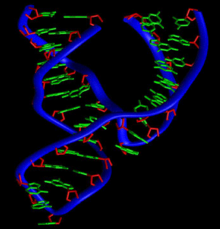
 Un bucle en forquita formado por unha molecula (monocatenaria) de
pre-ARNm
dobrada sobre si mesma. Estan salientadas as
nucleobases
(verdes) e a cadea ribosa-fosfato (azul).
Un bucle en forquita formado por unha molecula (monocatenaria) de
pre-ARNm
dobrada sobre si mesma. Estan salientadas as
nucleobases
(verdes) e a cadea ribosa-fosfato (azul).
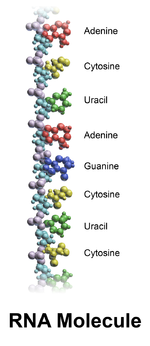 Bases dunha molecula de ARN.
Bases dunha molecula de ARN.
O
acido ribonucleico
ou
ARN
(
RNA
nas suas siglas en ingles) e un tipo de
acido nucleico
, unha molecula
polimerica
lineal formada por unidades menores chamadas
nucleotidos
. Interven en varias funcions bioloxicas importantes como a
codificacion xenetica
, e a descodificacion durante a
traducion de proteinas
, regulacion e expresion dos xenes. E unha das
macromoleculas
esenciais para a vida, xunto co
ADN
, as
proteinas
, os
lipidos
e os
carbohidratos
. Igual que o ADN, o ARN esta formado por unha cadea de nucleotidos, pero a diferenza do ADN, que forma unha dobre helice bicatenaria, a maioria dos ARNs son monocatenarios, ainda que se poden pregar sobre si mesmos. Os organismos celulares usan o
ARN mensaxeiro
(ARNm) para levar ao
ribosoma
a informacion xenetica (utilizando a secuencia das
bases
G, A, U, e C que significan
guanina
,
adenina
,
uracilo
e
citosina
), onde dirixira a sintese de proteinas especificas. A base uracilo e caracteristica do ARN (o ADN no seu lugar ten
timina
). Os nucleotidos do ARN levan o azucre
ribosa
, o que lle da o seu nome (
ribo
sa >
ribo
nucleico), a diferenza do ADN que leva
desoxirribosa
. O ARN
transcribese
a partir do ADN pola accion de
encimas
chamados
ARN polimerases
.
Algunhas moleculas de ARN tenen un papel moi activo nas celulas, xa que poden catalizar reaccions bioloxicas, controlar a
expresion xenica
, ou percibir e comunicar respostas a sinais celulares. Un destes activos procesos e a
sintese de proteinas
, unha funcion universal fundamental na que intervenen varios tipos de ARN: o ARNm leva a informacion de como ten que ser a secuencia da proteina, o
ARNt
leva os
aminoacidos
necesarios, e o
ARNr
e parte constituinte do
organulo
onde se realiza a sintese, o
ribosoma
, e ten unha actividade catalitica que une os aminoacidos entre si. Os
ribozimas
son ARNs con funcion encimatica. Moitos
virus
codifican a sua informacion xenetica nun
xenoma
de ARN.
 Representacion tridimensional da subunidade ribosomica de 50S. O ARN esta representado en ocre, as proteinas en azul. O sitio activo esta no medio (en vermello).
Representacion tridimensional da subunidade ribosomica de 50S. O ARN esta representado en ocre, as proteinas en azul. O sitio activo esta no medio (en vermello).
A estrutura quimica do ARN e moi similar a do
ADN
, pero diferenciase en cinco puntos principais:
- Numero de cadeas
. A diferenza do ADN, que normalmente e bicatenario, o ARN normalmente e monocatenario na maioria das suas funcions bioloxicas. Poren, o ARN pode, por apareamento de bases complementarias, formar dobres helices nunha mesma febra, como no
ARNt
. Hai tamen alguns ARN bicatenarios (en
virus
).
- Lonxitude
. O ARN e unha molecula mais curta: de decenas (ARNt) a miles de pares de bases (ARNm), mentres que o ADN nuclear ten millons.
- Azucre
. En vez do azucre
desoxirribosa
que ten o ADN, o ARN conten
ribosa
, a cal ten un grupo OH unido ao carbono 2', que non existe na desoxirribosa. O grupo 2'-hidroxilo da ribosa fai que o ARN sexa menos estable que o ADN porque e mais proclive a
hidrolise
.
 O grupo 2′-OH fai ao ARN mais sensible a hidrolise alcalina. A presenza de dous oxixenos en
cis
nas posicions 2′ e 3′ fai posible a ciclacion do fosfato nas posicions 2′ e 3′ cando se perde o proton do 2′-OH.
[
1
]
O grupo 2′-OH fai ao ARN mais sensible a hidrolise alcalina. A presenza de dous oxixenos en
cis
nas posicions 2′ e 3′ fai posible a ciclacion do fosfato nas posicions 2′ e 3′ cando se perde o proton do 2′-OH.
[
1
]
- Bases
. O ADN ten a
base nitroxenada
timina
, que e a complementaria da
adenina
, pero o ARN non ten timina senon
uracilo
, que funciona como base complementaria da adenina. O uracilo e unha forma
non metilada
da timina.
[
2
]
A sintese de uracilo e menos custosa enerxeticamente para os organismos vivos que a timina, xa que necesita unha etapa de sintese menos, que e a metilacion pola timidilato sintase. A presenza de timina no ADN permite que a celula detecte as lesions espontaneas da citosina, que e sensible a oxidacion. A
desaminacion
espontanea da citosina en presenza de oxixeno converte esta ultima en uracilo. A presenza de U no ADN e anormal, porque nel a base complementaria de A e T. Grazas a esta distincion entre a timina e o uracilo por medio do grupo metilo, o sistema de
reparacion por escision de base
pode detectar e corrixir o defecto no ADN. No ARN a desaminacion das citosinas produce uracilos e non e reparada. O ARN ten unha duracion moito mais curta que o ADN, xa que e degradado como media en 1 minuto e reciclado.
 O
uracilo
e a
base nitroxenada
caracteristica do ARN.
O
uracilo
e a
base nitroxenada
caracteristica do ARN.
- Estrutura
. O ARN non ten a estrutura en
dobre helice
do ADN. Igual que o ADN, a maioria dos ARNs bioloxicamente activos, como o
ARNm
,
ARNt
,
ARNr
,
ARN nuclear pequeno
, e outros
ARNs non codificantes
, contenen secuencias autocomplementarias que permiten que partes dunha mesma molecula de ARN se preguen
[
3
]
e se apareen entre elas para formar tramos de
dobre helice
. As analises destes ARNs revelaron que estan moi estruturados. A diferenza do ADN, as suas estruturas non consisten en longas
dobres helices
, senon en conxuntos de helices curtas empaquetadas xuntas en estruturas que lembran as das proteinas. Desta maneira o ARN pode conseguir realizar
catalises
quimicas, similares as dos
encimas
proteicos.
[
4
]
Por exemplo, a determinacion da estrutura do
ribosoma
, un complexo con actividades encimaticas que cataliza a formacion de
enlaces peptidicos
, revelou que o seu
sitio activo
esta composto enteiramente por ARN, a pesar de que no ribosoma hai tamen moitas proteinas asociadas.
[
5
]
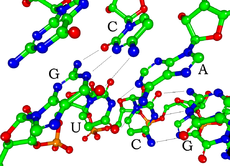 Os
pares de bases de Watson e Crick
do
ARN interferente pequeno
(non se mostran os atomos de hidroxeno).
Os
pares de bases de Watson e Crick
do
ARN interferente pequeno
(non se mostran os atomos de hidroxeno).
Cada
nucleotido
do ARN conten un azucre
ribosa
, no que os carbonos se numeran do 1' ao 5', unha base unida a posicion 1', en xeral,
adenina
(A),
citosina
(C),
guanina
(G), ou
uracilo
(U) (as veces aparecen bases especiais), e un fosfato. A adenina e a guanina son
purinas
, e a citosina e o uracilo son
pirimidinas
. Completase o
ribonucleotido
cun grupo
fosfato
unido a psoicion 3' dunha ribosa e a posicion 5' da seguinte, establecendo un
enlace fosfodiester
. Os grupos fosfato tenen unha carga negativa a
pH
fisioloxico, facendo que o ARN sexa unha molecula cargada (polianion). As bases forman
enlaces de hidroxeno
entre as bases enfrontadas citosina e guanina, e entre a adenina e o uracilo, e as veces o apareamento
cambaleante
entre a guanina e o uracilo.
[
6
]
Poren, son posibles outras interaccions, como que un grupo de bases adeninas se unan entre si nunha protuberancia,
[
7
]
ou o
tetrabucle
GNRA, que ten o apareamento guanina?adenina.
[
6
]
 Estrutura quimica do ARN.
Estrutura quimica do ARN.
Unha importante caracteristica estrutural do ARN que o distingue do ADN e a presenza dun grupo
hidroxilo
na posicion 2' do azucre ribosa. A presenza deste grupo funcional causa que a helice adopte a
xeometria da forma A
en vez da da
forma B
, que e a que normalmente se observa no ADN.
[
8
]
Isto orixina a formacion dun suco maior moi profundo e estreito e un suco menor largo e pouco profundo.
[
9
]
Unha segunda consecuencia da presenza do grupo 2'-hidroxilo e que en rexions flexibles conformacionalmente dunha molecula de ARN (e dicir, non implicadas na formacion da dobre helice), este pode atacar quimicamente o enlace fosfodiester adxacente e cortar a molecula.
[
10
]
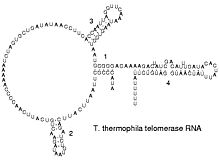 Estrutura secundaria
dun
ARN de telomerase
.
Estrutura secundaria
dun
ARN de telomerase
.
Para transcribir o ARN utilizanse so catro bases (adenina, citosina, guanina e uracilo),
[
11
]
pero estas bases e os azucres unidos a elas poden ser modificados de numerosas maneiras durante a maduracion dos ARNs. A
pseudouridina
(Ψ), na cal o enlace entre o uracilo e a ribosa cambia dun enlace C?N a un enlace C?C, e a ribotimidina (T, unha timina unida a ribosa) poden encontrarse en varios sitios nos ARNs (o mais notable e o bucle TΨC dos
ARNts
).
[
12
]
Outra base modificada importante e a
hipoxantina
, que e unha adenina desaminada cuxo
nucleosido
se chama
inosina
(I). A inosina xoga un papel clave na
hipotese do cambaleo
do
codigo xenetico
.
[
13
]
Hai mais de 100 nucleosidos modificados que aparecen na natureza,
[
14
]
A maior diversidade estrutural de modificacions atopase no ARNt,
[
15
]
mentres que a pseudouridina e os nucleosidos con
2'-O-metilribosa
estan a miudo presentes no
ARNr
.
[
16
]
As funcions especificas de moitas destas modificacions do ARN non se comprenden totalmente. Con todo, e importante que, no ARN ribosomico, moitas das
modificacions postranscricionais
ocorren en rexions altamente funcionais, como no centro da
peptidil transferase
e a interface entre as subunidades, o que implica que son importantes para o funcionamento normal.
[
17
]
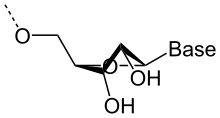 Conformacion C2′-endo, observada nas helices de tipo B do ADN.
Conformacion C2′-endo, observada nas helices de tipo B do ADN.
 Conformacion C3′-endo, observada nas helices de tipo A do ARN.
Conformacion C3′-endo, observada nas helices de tipo A do ARN.
A presenza do OH no carbono 2' da ribosa orixina diferenzas estereoquimics con respecto ao ARN. Poden formarse dous
conformeros
principais do azucre, chamados C2′-endo e C3′-endo. No ARN, ao levar un atomo de oxixeno no carbono 2', a posicion privilexiada e a 3'-endo,
[
18
]
, o que modifica profundamente a estrutura das dobres helices entre febras de ARN nos casos en que estas se forman. Estes duplex de ARN forman unha helice de tipo A, diferente da observada normalmente no ADN, que e de tipo B, na que a
desoxirribosa
esta en conformacion C2′-endo
[
19
]
.
O
enantiomero
que aparece na natureza do ARN e o
D
-ARN composto de
D
-ribonucleotidos. Todos os
centros quirais
estan localizados na
D
-ribosa. Non obstante, pode sintetizarse
L
-ARN utilizando
L
-ribosa ou mais ben
L
-ribonucleotidos. O
L
-ARN e moito mais estable ante a degradacion por
RNase
.
[
20
]
 Estrutura "en forquita", ou
talo-bucle
, formada por unha
secuencia palindromica
no ARN.
Estrutura "en forquita", ou
talo-bucle
, formada por unha
secuencia palindromica
no ARN.
A
estrutura secundaria
dun ARN e a descricion do conxunto de apareamentos de bases internos dentro dunha mesma molecula monocatenaria de ARN.
[
21
]
. Este conxunto de apareamentos inducido por unha topoloxia particular, esta composto de rexions en helice (talos) e rexions non apareadas (bucles). Por extension, a estrutura secundaria comprende tamen a descricion desta topoloxia.
A formacion de estruturas secundarias nun ARN monocatenario orixinase pola existencia de rexions que contenen
secuencias palindromicas
, que poden aparearse para formar localmente unha estrutura en dobre helice. Por exemplo, se o ARN conten as duas secuencias seguintes : --GUGCCACG------CGUGGCAC--, estas forman unha secuencia palindromica, porque os nucleotidos do segundo segmento son complementarios dos do primeiro, despois da inversion do seu sentido de lectura ; estes dous segmentos poden enton aparearse de maneira
antiparalela
para formar unha rexion localmente en duplex. A rexion entre os dous segmentos forma un bucle que une as duas febras apareadas, e a zona apareada forma un talo. Falase enton de estrutura en forquita ou en
talo-bucle
.
 Topoloxia das diferentes estruturas secundarias que se poden atopar no ARN.
Topoloxia das diferentes estruturas secundarias que se poden atopar no ARN.
Nos ARNs de maior lonxitude, poden existir estruturas mais complexas que se orixinan polo apareamento de varaias rexions complementarias ou secuencias palindromicas. En funcion do xeito no que son "encaixadas" estas diferentes rexions, obtenense elementos topoloxicos variados, con talos de rexions apareadas e diversos tipos de bucles, como son:
[
22
]
- bucles terminais
, situados no extremo dun talo ;
- bucles internos
, que conectan dous talos ;
- bucles multiples
, que conectan tres talos ou mais e constituen os puntos de ramificacion da estrutura ;
- protuberancias
ou
bucles laterais
, que estan situados sobre unha soa das febras da helice. A continuidade da helice en xeral non esta afectada e as bases quedan colocadas unha enriba da outra de maneira coaxial, a un lado e outro da protuberancia.
Non sempre existe unha estrutura estable unica para unha secuencia dada e ocorre que certos ARN poden adoptar varias conformacions alternativas en funcion da union dun ligando (proteico ou pequena molecula) ou das condicions fisico-quimicas (
forza ionica
,
pH
). En xeral podese seguir a evolucion da formacion ou a "fusion" (separacion das febras) da estrutura secundaria dun ARN por medicions
espectroscopicas
. Por exemplo, a absorcion
ultravioleta
das bases do ARN e maior no estado "fundido" que en dobre helice (hipercromatismo).
[
23
]
 O ARNt e un ARN cunha estrutura complexa.
O ARNt e un ARN cunha estrutura complexa.
A forma funcional das moleculas de ARN monocatenario, igual que ocorre coas proteinas, con frecuencia require que adopten unha estrutura terciaria especifica. O armazon para esta estrutura proporcionano elementos da estrutura secundaria que son os enlaces de hidroxeno dentro da molecula. Isto orixina varios "dominios" estruturais reconecibles de estrutura secundaria como
bucles en forquita
, protuberancias, e
bucles internos
.
[
24
]
Como o ARN e unha molecula cargada, son necesarios ions metalicos como o
Mg
2+
para estabilizar moitas
estruturas secundarias
e
terciarias
.
[
25
]
Os apareamentos canonicos ou non canonicos poden intervir entre rexions distantes da estrutura secundaria, localizadas a miudo nos bucles, o que permite estabilizar un pregsamento compacto da estrutura. Ademais dos
apareamentos de Watson e Crick
canonicos, poden atoparse tamen apareamentos de
Hoogsteen
[
26
]
e outros.
[
27
]
.
Entre as interaccions non canonicas a gran distancia estan:
- pseudonos
, estruturas formadas pola interaccion dun bucle cunha rexion situada no exterior do talo que a delimita;
[
28
]
- triplex
(de tres febras), que se orixinan cando unha rexion de febra simple se nsire no suco maior dunha rexion en helice ;
- interaccions
tetrabucle
-receptor
: interaccions entre bucles hiperestables de catro nucleotidos (tetrabucles) e estruturas en duplex ou quasi-duplex.
[
29
]
 Comparacion das estruturas da helice B do ADN e da helice A do ARN. O suco maior esta modificado.
[
30
]
[
31
]
Comparacion das estruturas da helice B do ADN e da helice A do ARN. O suco maior esta modificado.
[
30
]
[
31
]
A helice que adopta o ARN naqueles casos en que forma un duplex non e a de tipo B do ADN, senon unha helice de tipo A, que ten propiedades xeometricas bastante diferentes. O numero de pares de bases no paso de rosca da helice e de 11 (no
ADN B
son 10). O plano dos pares de bases non e perpendicular ao eixe da helice, senon que forma un angulo duns 75° con este.
[
32
]
,
[
33
]
Isto da lugar a un desprazamento do eixe da helice que xa non pasa polo centro do apareamento de bases, senon polo interior do suco maior. Isto induce un aumento do diametro da helice que pasa de ter uns 20 A no ADN B a uns 26 A na forma A do ARN.
[
34
]
A xeometria dos dous sucos esta profundamente afectada: o suco menor faise mais accesible, mentres que o suco maior faise mais profundo e estreito. Isto ten un impacto sobre a maneira na que o ARN duplex pode interaccionar coas proteinas, porque a estreitura do suco maior e unha barreira a accesibilidade dos
ligandos
proteicos.
[
35
]
.
O material xenetico das celulas encontrase en forma de
ADN
. Dentro das moleculas de ADN encontrase a informacion necesaria para sintetizar as proteinas que utiliza o organismo; pero o ADN non se
traduce
directamente en
proteinas
, senon que o fai por intermediacion do ARNm.
Nas
celulas eucariotas
o ADN encontrase encerrado no
nucleo
. A sintese de ADN faise no nucleo, asi como tamen a sintese de ARN (tamen en
mitocondrias
e
cloroplastos
), pero a sintese de proteinas acontece no
citoplasma
. O mecanismo polo cal a informacion se transvasa dende o nucleo celular ao citoplasma e mediante a trascricion do ARN a partir do ADN, a maduracion do ARN no nucleo (na que sofre modificacions), o seu transporte ao citoplasma e a traducion de proteinas nos
ribosomas
do citoplasma a partir de ARN.
Durante este fluxo de informacion xenetica producense apareamentos complementarios de bases entre o ARN e o ADN durante a transcricion, e durante a traducion entre os
codons
do ARNm e os
anticodons
do ARNt. Entre dous ARNs (ou entre partes apareadas dunha mesma molecula de ARN) os apareamentos normais son A-U, G-C. No caso da transcricion a complementariedade e:
- Uracilo
(U)
do ARN con
Adenina
(A) do ADN
- Adenina
(A)
do ARN con
Timina
(T)
do ADN
- Citosina
(C)
con
Guanina
(G)
(en ambos)
A sintese do ARN faise case sempre na natureza a partir dun molde de ADN, catalizada por un encima
ARN polimerase
, que usa o ADN como molde, nun proceso denominado
transcricion
. A iniciacion da transcricion empeza coa union do encima a secuencia do
promotor
presente no ADN (xeralmente "
augas arriba
" do xene). A
dobre helice
do ADN e desenrolada pola actividade de
helicase
do encima. O encima despois progresa ao longo da febra molde do ADN en
direccion 3’-5’
, sintetizando unha molecula de ARN complementaria que se elonga en direccion 5’-3’. Unha secuencia de ADN, o
terminador
, indica onde tera lugar a terminacion da sintese de ARN.
[
36
]
O transcrito primario de ARN xeralmente debe ser ulteriormente
modificado
por encimas despois da transcricion (ver a seguinte seccion).
Existen tamen varias
ARN polimerases ARN dependentes
que usan o ARN como o seu molde para a sintese dunha nova febra de ARN. Por exemplo, un numero de
virus de ARN
(como os
poliovirus
) usan este tipo de encima para replicar o seu material xenetico.
[
37
]
Ademais, a ARN polimerase ARN dependente forma parte da via de
interferencia de ARN
en moitos organismos.
[
38
]
 Eliminacion dos introns dun
pre-ARNm
durante o splicing.
Eliminacion dos introns dun
pre-ARNm
durante o splicing.
A maduracion dos ARN comprende un conxunto de
modificacions postranscricionais
principalmente observadas en eucariotas que son moi importantes para obter un ARN maduro funcional. Estas modificacions sofrenas os diversos tipos de ARN. No caso dos
pre-ARNm
as principais modificacions son a union dunha
carapucha 5'
nun dos extremos e dunha cola poliA (
poliadenilacion
) no 3′, e o
splicing
ou empalme na parte central, realizado polo
espliceosoma
, durante o que se eliminan os
introns
. Poden introducirse tamen modificacions quimicas ao nivel da base ou da ribosa e pode haber unha
edicion do ARN
.
Nas bacterias e en certas mitocondrias, a poliadenilacion dos ARN e, ao contrario, un sinal para a degradacion.
[
39
]
.
Despois da sua transcricion poloa ARN polimerase, certos ARN sofren modificacions quimicas pola accion de encimas especificos. Os principais ARNs que sofren estas modificacions son os
ARNt
e os
ARNr
. Poderiase considerar que as
metilacions
que se realizan durante a formacion da
carapucha 5'
son tamen modificacions de
nucleotidos
particulares. As modificacions poden producirse nas bases ou na ribosa. As principais modificacions que se producen son as seguintes:
Nos ARNt, a introducion de nucleotidos modificados contribue a aumentar a estabilidade das moleculas
[
41
]
.
A
edicion dos ARN
consiste nunha modificacion da secuencia do ARN posterior a transcricion. Despois da edicion a secuencia do ARN e diferente da (que corresponderia por complementariedade) do ADN. A edicion pode consistir en insercions, delecions, e substitucions de bases de nucleotidos na molecula de ARN. Estas modificacions son efectuadas por encimas que actuan sobre o ARN.
 Estrutura do
ribozima cabeza de martelo
, un
ribozima
que corta o ARN.
Estrutura do
ribozima cabeza de martelo
, un
ribozima
que corta o ARN.
O ARN mensaxeiro (ARNm) e o ARN que leva a informacion do ADN ao
ribosoma
, aos sitios de sintese de proteinas (
traducion
) da celula (
ribosomas
). A secuencia codificante formada polos
codons
do ARNm determina a secuencia de
aminoacidos
da
proteina
que se vai producir.
[
42
]
Poren, moitos ARNs non codifican proteinas (un 97% dos ARNs transcritos son
ARN non codificante
en eucariotas
[
43
]
[
44
]
[
45
]
[
46
]
).
Este
ARN non codificante
("ARNnc") pode estar directamente codificado polos seus propios xenes (xenes de ARN), pero pode tamen derivar de
introns
que formaban parte de ARNms.
[
47
]
Os exemplos mais importantes de ARNs non codficantes son o
ARN transferente
(ARNt) e o
ARN ribosomico
(ARNr), que estan ambos implicados no proceso de traducion de proteinas.
[
2
]
Hai tamen ARNs non codificantes implicados na
regulacion xenica
, o
procesamento do ARN
e outros papeis. Certos ARNs poden
catalizar
reaccions quimicas como cortar e
ligar
outras moleculas de ARN,
[
48
]
e catalizar a formacion de
enlaces peptidicos
no
ribosoma
,
[
5
]
o que e unha actividade similar a dos encimas proteicos; estes ARN denominanse
ribozimas
.
-
O
ARN mensaxeiro
(ARNm) leva informacion sobre a secuencia dunha proteina aos
ribosomas
, que son as fabricas onde se realiza a sintese proteica na celula. O ARNm esta
codificado
de modo que cada tres nucleotidos do ARN (o que se chama
codon
) corresponda a un aminoacido da proteina. Nas celulas eucariotas, un ARNm precursor (
pre-ARNm
) que foi transcrito do ADN, e
procesado
orixinando un ARNm maduro. Durante esta maduracion eliminanse os
introns
, que son segmentos non codificantes que estan intercalados na molecula do pre-ARNm. Durante a maduracion tamen se modifican os dous extremos do ARNm (
poliadenilacion
e adicion da
carapucha 5'
). O ARNm e despois exportado desde o
nucleo
ao
citoplasma
, onde se une a un ribosoma e e
traducido
na sua proteina correspondente coa axuda do
ARNt
. En
celulas procariotas
, que carecen de nucleo, o ARNm pode unirse por un extremo aos ribosomas antes de que acabe a sua transcricion do ADN polo outro extremo. Despois de pasado un tempo a mensaxe (o ARN) degradase liberando os
nucleotidos
que o componen coa axuda de encimas
ribonucleases
.
[
42
]
O
ARN de transferencia
(ARNt) e unha pequena cadea de ARN duns 80
nucleotidos
que transporta un aminoacido especifico a unha cadea polipeptidica en crecemento no sitio ribosomico de sintese proteica durante a traducion. Ten unha estrutura con catro brazos (en
folla de trevo
), que estan pregados uns sobre os outros, e ten sitios para a union do aminoacido e unha rexion
anticodon
para o reconecemento do
codon
do ARN (ambos os dous son complementarios en bases e unense por enlaces de hidroxeno).
[
47
]
O
ARN ribosomico
(ARNr) forma parte do ribosoma, xunto con diversas proteinas, e e o componente catalitico do ribosoma. Os ribosomas eucariotas contenen catro ARNrs distintos clasificados polo seu
coeficiente de sedimentacion
(S): ARNr de
18S
,
5,8S
,
28S
e
5S
. Tres destes ARNr sintetizanse no
nucleolo
, e un sintetizase noutras partes do nucleo. No citoplasma o ARNr e diversas proteinas combinanse para formar dous complexos ribonucleoproteicos que son as duas subunidades do ribosoma, que se unen formando un ribosoma funcional. O ribosoma unese ao ARNm e leva a cabo a sintese de proteinas. Poden unirse varios ribosomas a vez a unha soa molecula de ARNm, formando un
polisoma
.
[
42
]
A gran maioria do ARN que se encontra nunha celula eucariota tipica e ARNr.
En moitas
bacterias
e
plastos
atopase un ARN especial chamado
ARN transferente-mensaxeiro
(ARNtm). Este ARN etiqueta as proteinas codificadas por ARNms que carecen de
codons de parada
para que sexan degradados e impide que o ribosoma quede atascado.
[
49
]
Varios tipos de ARN poden regular a baixa a
expresion xenica
ao ser complementarios dunha parte dun ARNm ou dun xene do ADN. Os
microARNs
(miARN; de 21-22
nt
) atopanse en eucariotas e actuan por medio de
interferencia de ARN
(RNAi), na que un complexo efector de miARN e encimas poden clivar un ARNm complementario, bloquear a traducion do ARNm, ou acelerar a sua degradacion.
[
50
]
[
51
]
Ainda que a miudo se producen
ARNs interferentes pequenos
(siRNA; de 20-25 nt) pola degradacion do ARN viral, hai tamen fontes endoxenas destes siRNAs, que son producidos de forma natural normal pola celula.
[
52
]
[
53
]
Os ARN interferentes pequenos actuan por medio de interferencia de ARN dun modo similar aos miARNs. Alguns miARNs e siRNAs poden causar que os seus xenes diana sexan
metilados
, facendo que asi
diminua
ou
aumente
a transcricion de ditos xenes.
[
54
]
[
55
]
[
56
]
Os animais tenen tamen
ARNs que interaccionan con piwi
(piRNA; de 29-30 nt) que son activos nas celulas da
lina xerminal
e crese que son unha defensa contra os
transposons
e xogan un papel na
gametoxenese
.
[
57
]
[
58
]
Moitos procariotas tenen ARNs
CRISPR
, un sistema regulatorio similar a interferencia de ARN.
[
59
]
Os
ARNs antisentido
estan moi estendidos; a maioria regulan a baixa un xene, pero uns poucos son activadores da transcricion.
[
60
]
Un modo en que poden actuar os ARN antisentido e mediante a sua union ao ARNm, formando un ARN de dobre cadea, que e degradado encimaticamente.
[
61
]
Hai moitos
ARNs non codificantes longos
que regulan xenes en eucariotas,
[
62
]
un deles e
Xist
, que recobre un dos
cromosomas X
nas femias de mamifero e o
inactiva
.
[
63
]
Un ARNm pode conter na propia molecula elementos regulatorios, como
riboswitches
, nas suas rexions non traducidas
5'UTR
ou
3'UTR
; estes
elementos cis reguladores
regulan a actividade do ARNm.
[
64
]
As rexions non traducidas (UTR) poden conter tamen elementos que regulan outros xenes.
[
65
]
 Unha modificacion comun no ARN e o cambio de uridina a pseudouridina.
Unha modificacion comun no ARN e o cambio de uridina a pseudouridina.
Moitos ARNs tenen a funcion de modificar outros ARNs.
Os ARNm eucariotas tenen
introns
antes da sua maduracion. Os introns son eliminados por
splicing
dos
pre-ARNm
polos
espliceosomas
, que contenen varios
ARNs nucleares pequenos
(snRNA),
[
2
]
ou outras veces os introns poden ser
ribozimas
que realizan o splicing eles mesmos.
[
66
]
O ARN pode tamen ser alterado modificando os seus nucleotidos a outros distintos dos habituais
A
,
C
,
G
e
U
.
En eucariotas, as modificacions dos nucleotidos do ARN son en xeral dirixidas por
ARNs nucleolares pequenos
(snoRNA; 60-300 nt),
[
47
]
que se encontran no
nucleolo
e nos
corpos de Cajal
. Os ARNs nucleolares pequenos asocianse con encimas e guianos a un lugar no ARN e unense por apareamento de bases con el. Estes encimas despois realizan a modificacion do nucleotido. Os ARN ribosomicos e ARN transferentes son amplamente modificados, pero os ARN nucleares pequenos e os ARN mensaxeiros poden tamen sufrir este tipo de modificacion de bases.
[
67
]
[
68
]
O ARN tamen pode ser
metilado
.
[
69
]
[
70
]
Mencionamos no apartado anterior que varios tipos de ARN que intervenen no splicing ou na interferencia poden guiar moleculas a zonas do ADN ou doutros ARNs especificas coas que son complementarios. Ademais dos mencionados, nesta funcion de guia estan tamen:
- TERC
(
componente de ARN da telomerase
), que e a subunidade de ARN do encima
telomerase
: este ARN estruturado esta asociado ao encima de tipo
transcriptase inversa
que sintetiza os
telomeros
(extremos dos cromosomas). Conten unha secuencia que serve de substrato a telomerase para sintetizar o ADN telomerico de secuencia complementaria.
[
71
]
. Por tanto, guia a actividade do encima, e ademais serve de molde (en lugar de aparearse co substrato).
- lincARN
, que son grandes ARNs interxenicos non codificantes presentes nos mamiferos, que son transcritos (igual que o ARNm) pola
ARN polimerase II
. A sua gran lonxitude permitelles adoptar unha estrutura tridimensional complexa. Estas estruturas permiten a sua interaccion con diferentes cofactores transcricionais como o
hnRNP-K
ou o
PRC2
(principalmente inhibidores da transcricion). Estes complexos son despois guiados grazas aos lincARN as secuencias reguladoras dos xenes para inhibir a sua expresion. A ligazon dos lincARN co ADN implica un apareamento de bases ARN-ADN despois de que se abra a dobre helice do ADN.
[
72
]
.
 Estrutura da
ribonuclease P
, un
encima
presente en todas as celulas vivas cuxa actividade encimatica depende dun ARN.
Estrutura da
ribonuclease P
, un
encima
presente en todas as celulas vivas cuxa actividade encimatica depende dun ARN.
Desde a decada de 1980 sabese que o ARN pode ter capacidades encimaticas. Os primeiro descubrironos
Thomas Cech
e
Sidney Altman
estudando o
protozoo
Tetrahymena
(que ten un intron con autosplicing) e a
ribonuclease P
(que interven na maduracion do ARNt), respectivamente, polo que foron recompensados co
premio Nobel de Quimica
de 1989.
[
73
]
[
74
]
Nestes dous casos o ARN por si so pode catalizar unha reaccion de clivaxe (corte) ou de
transesterificacion
especifica en ausencia de
proteina
, do mesmo modo que o faria un encima proteico. Estes ARNs cataliticos denominanse
ribozimas
. En xeral, a actividade dos ribozimas depende da sua estrutura tridimensional especifica, mediante a cal poden reconecer o substrato e catalizar a reaccion nunha zona da molecula.
 Estrutura do
ribozima cabeza de martelo
presente no xenoma dun
viroide
que infecta a planta do
aguacate
.
Estrutura do
ribozima cabeza de martelo
presente no xenoma dun
viroide
que infecta a planta do
aguacate
.
Posteriormente descubrironse outros ribozimas naturais, como: ribozimas de
viroides
e
virus satelites
(autoclivaxe),
[
75
]
o ribosoma (na sua actividade de peptidil-transferase e de descodificacion, que ocorren en zonas formadas so por ARN),
[
76
]
o
espliceosoma
(cataliza o splicing do ARNm porque ten actividade riboenzimatica),
[
77
]
certos
riboswitches
(rexions dos ARNm con capacidade de cortar moleculas en presenza dun ligando),
[
78
]
, e mesmo hai ribozimas sinteticos (obtidos pola tecnica
SELEX
, chamados
aptameros
, que poden realizar unha reaccion determinada desexada).
[
79
]
Igual que fai o ADN, o ARN tamen pode almacenar informacion xenetica. Os
virus de ARN
tenen
xenomas
compostos de ARN que codifica varias proteinas. Algunhas destas proteinas replican o xenoma viral, mentres que outras protexen o xenoma (cunha
capside
) cando a particula virica se move dunha celula hospede a outra. Os
viroides
son outro grupo de patoxenos de plantas parecidos a virus, pero que consisten so en ARN (carecen de capside proteica), que non codifican ningunha proteina e son replicados por unha
polimerase
da celula hospede da planta.
[
80
]
Os virus con
reversotranscricion
replican os seus xenomas producindo copias de ADN reversotranscribindo o seu xenoma de ARN; estas copias de ADN son despois transcritas a un novo ARN. Os
retrotransposons
tamen se espallan ao copiaren ADN e ARN un do outro,
[
81
]
e a
telomerase
conten un ARN que se utiliza como molde para construir os extremos dos
cromosomas
eucariotas.
[
82
]
Os ARN bicatenarios (dsRNA) son ARNs con duas febras complementarias, similares ao ADN que se encontra en todas as celulas. Os ARNs bicatenarios forman o material xenetico dalguns virus (
virus de ARN bicatenario
). O ARN bicatenario como o de certos ARNs virais ou o
ARN interferente pequeno
poden desencadear a
interferencia de ARN
en eucariotas, e a resposta ao
interferon
en
vertebrados
.
[
83
]
[
84
]
[
85
]
[
86
]
Usos terapeuticos e biotecnoloxicos
[
editar
|
editar a fonte
]
O ARN e utilizado hoxe nun certo numero de aplicacions en
bioloxia molecular
, en especial grazas ao proceso da
interferencia de ARN
, que consiste na introducion nas celulas eucariotas de curtos fragmentos de ARN bicatenario de
ARN interferente pequeno
, dunha lonxitude duns 20 pares de bases. Estes ARNs van ser utilizados por unha maquinaria celular que pode degradar os ARNm de maneira especifica. So seran degradados os ARNm que contenan unha secuencia correspondente a do ARN interferente pequeno, o que fai diminuir selectivamente a expresion dunha determinada proteina (a codificada no ARNm).
[
87
]
. Este enfoque tecnoloxico e mais simple e rapido que o establecemento de linas de ratos con xenes inactivados (
knock-out
) e denominase
knock-down
.
Prevese o uso desta tecnica con fins terapeuticos, por exemplo dirixindo os ARNs contra os xenes virais para loitar contra as infeccions, ou os
oncoxenes
, no caso de cancros.
[
88
]
. Poren, compre que estes ARN interferentes pequenos sexan estabilizados para evitar a sua degradacion polas
ribonucleases
e dirixir a sua accion contra as celulas diana concernidas.
 A esquerda, Robert W. Holley xunto co seu equipo de investigacion.
A esquerda, Robert W. Holley xunto co seu equipo de investigacion.
A investigacion sobre o ARN levou a facer moitos importantes descubrimentos bioloxicos e mereceu varios
premios Nobel
. Os
acidos nucleicos
foron descubertos en 1868 por
Friedrich Miescher
, que chamou a este material "nucleina", xa que o atopou no
nucleo celular
.
[
89
]
Descubriuse despois que as
celulas procariotas
, que carecen de nucleo, tamen continan acidos nucleicos. Sospeitabase que o ARN xogaba un papel na sintese de proteinas xa en 1939.
[
90
]
Severo Ochoa
ganou en 1959 o
Premio Nobel de Medicina
(compartido con
Arthur Kornberg
) polo seu descubrimento dun
encima
que pode sintetizar ARN no laboratorio.
[
91
]
Poren, o encima descuberto por Ochoa (a
polinucleotido fosforilase
) viuse mais tarde que era responsable da degradacion do ARN nas celulas, e non da sua sintese. En 1956 Alex Rich e David Davies hibridaron duas febras separadas de ARN para formar o primeiro cristal obtido de ARN cuxa estrutura podia determinarse por
cristalografia de raios X
.
[
92
]
En 1965,
Robert W. Holley
obtivo a secuencia de 77 nucleotidos dun ARNt de
levedo
,
[
93
]
polo que recibiu en 1968 o Premio Nobel de Medicina (compartido con
Har Gobind Khorana
e
Marshall Nirenberg
).
En 1967,
Carl Woese
hipotetizou que o ARN poderia ser catalitico e suxeriu que as formas de vida mais primordiais da Terra primitiva (moleculas autorreplicantes) poderian depender do ARN tanto para almacenar a informacion xenetica coma para catalizar reaccions bioquimicas, o que se chama
hipotese do mundo de ARN
.
[
94
]
[
95
]
A inicios da decada de 1970, descubrironse os
retrovirus
e a
transcriptase inversa
, demostrando por primeira vez que os encimas podian facer copias de ADN a partir dun molde de ARN (o contrario do fluxo de transmision da informacion xenetica normal). Por estes traballos,
David Baltimore
,
Renato Dulbecco
e
Howard Temin
recibiron o Premio Nobel en 1975.
En 1976,
Walter Fiers
e o seu equipo determinaron a primeira secuencia nucleotidica completa dun xenoma de
virus de ARN
, que era o do
bacteriofago MS2
.
[
96
]
En 1977, descubrironse os
introns
e o
splicing
de ARN en virus de mamiferos e en xenes celulares, o que supuxo que en 1993
Philip Sharp
e
Richard Roberts
fosen galardoados co Premio Nobel.
As moleculas de ARN cataliticas (
ribozimas
) foron descubertas a inicios da decada de 1980, polo cal recibiron o Premio Nobel de 1989
Thomas Cech
e
Sidney Altman
. En 1990, descubriuse que en
Petunia
xenes introducidos podian silenciar xenes similares da propia planta, o que hoxe sabemos se debia a
interferencia de ARN
.
[
97
]
[
98
]
A aproximadamente o mesmo tempo, atoparonse uns ARNs de 22 nt de longo, hoxe chamados
microARNs
, que tinan un papel no desenvolvemento de
Caenorhabditis elegans
.
[
99
]
Os estudos sobre a interferencia de ARN supuxeron o Premio Nobel para
Andrew Fire
e
Craig Mello
en 2006, e concedeuse outro Nobel polos estudos sobre transcricion do ARN a
Roger Kornberg
no mesmo ano. O descubrimento de ARNs reguladores de xenes levou a intentar desenvolver
farmacos
feitos de ARN, como os derivados de certos
ARN interferentes pequenos
, para silenciar xenes.
[
100
]
En marzo de 2015, obtiveronse no laboratorio compostos organicos que forman parte do ADN e ARN basicos para a
vida
, como o
uracilo
,
citosina
e
timina
, en condicions que simulaban as do
espazo exterior
, usando compostos de partida como
pirimidina
, que se encontrou en
meteoritos
. A pirimidina, igual que os
hidrocarburos aromaticos policiclicos
, os compostos mais ricos en carbono atopados no
Universo
, puideron formarse nas estrelas
xigantes vermellas
ou no
po cosmico
interestelar e nubes de gas.
[
101
]
A
hipotese do mundo de ARN
e unha
hipotese
segundo a cal o ARN seria a primeira molecula que apareceu na Terra con capacidade de almacenar informacion xenetica e ter actividade catalitica, funcions nas que mais tarde se especializarian o ADN e as proteinas. Esta hipotese permite unha explicacion da aparicion das diferentes funcions bioloxicas no estudo da
orixe da vida
.
- ↑
Markham R., Smith J.D. (1952).
"The Structure of Ribonucleic Acids 1. Cyclic nucleotides produced by ribonuclease and by alkaline hydrolysis"
.
Biochemical Journal
(en
ingles
)
52
(4): 552?557.
PMID
13018277
.
- ↑
2,0
2,1
2,2
Berg JM, Tymoczko JL, Stryer L (2002).
Biochemistry
(5th ed.). WH Freeman and Company. pp. 118?19, 781?808.
ISBN
0-7167-4684-0
.
OCLC
179705944
.
- ↑
I. Tinoco and C. Bustamante (1999). "How RNA folds".
J. Mol. Biol.
293
(2): 271?281.
PMID
10550208
.
doi
:
10.1006/jmbi.1999.3001
.
- ↑
Higgs PG (2000).
"RNA secondary structure: physical and computational aspects"
.
Quarterly Reviews of Biophysics
33
(3): 199?253.
PMID
11191843
.
doi
:
10.1017/S0033583500003620
.
- ↑
5,0
5,1
Nissen P, Hansen J, Ban N, Moore PB, Steitz TA; Hansen; Ban; Moore; Steitz (2000). "The structural basis of ribosome activity in peptide bond synthesis".
Science
289
(5481): 920?30.
Bibcode
:
2000Sci...289..920N
.
PMID
10937990
.
doi
:
10.1126/science.289.5481.920
.
- ↑
6,0
6,1
Lee JC, Gutell RR; Gutell (2004). "Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs".
J. Mol. Biol.
344
(5): 1225?49.
PMID
15561141
.
doi
:
10.1016/j.jmb.2004.09.072
.
- ↑
Barciszewski J, Frederic B, Clark C (1999).
RNA biochemistry and biotechnology
. Springer. pp. 73?87.
ISBN
0-7923-5862-7
.
OCLC
52403776
.
- ↑
Salazar M, Fedoroff OY, Miller JM, Ribeiro NS, Reid BR; Fedoroff; Miller; Ribeiro; Reid (1992). "The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution".
Biochemistry
32
(16): 4207?15.
PMID
7682844
.
doi
:
10.1021/bi00067a007
.
- ↑
Hermann T, Patel DJ; Patel (2000). "RNA bulges as architectural and recognition motifs".
Structure
8
(3): R47?R54.
PMID
10745015
.
doi
:
10.1016/S0969-2126(00)00110-6
.
- ↑
Mikkola S, Nurmi K, Yousefi-Salakdeh E, Stromberg R, Lonnberg H; Stenman; Nurmi; Yousefi-Salakdeh; Stromberg; Lonnberg (1999). "The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group".
Perkin transactions 2
(8): 1619?26.
doi
:
10.1039/a903691a
.
- ↑
Jankowski JAZ, Polak JM (1996).
Clinical gene analysis and manipulation: Tools, techniques and troubleshooting
. Cambridge University Press. p. 14.
ISBN
0-521-47896-0
.
OCLC
33838261
.
- ↑
Yu Q, Morrow CD; Morrow (2001).
"Identification of critical elements in the tRNA acceptor stem and TΨC loop necessary for human immunodeficiency virus type 1 infectivity"
.
J Virol.
75
(10): 4902?6.
PMC
114245
.
PMID
11312362
.
doi
:
10.1128/JVI.75.10.4902-4906.2001
.
- ↑
Elliott MS, Trewyn RW; Trewyn (1983). "Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine".
J. Biol. Chem.
259
(4): 2407?10.
PMID
6365911
.
- ↑
Cantara, WA; Crain, PF; Rozenski, J; McCloskey, JA; Harris, KA; Zhang, X; Vendeix, FA; Fabris, D; Agris, PF (January 2011).
"The RNA Modification Database, RNAMDB: 2011 update"
.
Nucleic Acids Research
39
(Database issue): D195?201.
PMC
3013656
.
PMID
21071406
.
doi
:
10.1093/nar/gkq1028
.
- ↑
Soll D, RajBhandary U (1995).
TRNA: Structure, biosynthesis, and function
. ASM Press. p.
165
.
ISBN
1-55581-073-X
.
OCLC
183036381
.
- ↑
Kiss T (2001).
"Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs"
.
The EMBO Journal
20
(14): 3617?22.
PMC
125535
.
PMID
11447102
.
doi
:
10.1093/emboj/20.14.3617
.
- ↑
King TH, Liu B, McCully RR, Fournier MJ; Liu; McCully; Fournier (2002). "Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center".
Molecular Cell
11
(2): 425?35.
PMID
12620230
.
doi
:
10.1016/S1097-2765(03)00040-6
.
- ↑
M. Sudaralingam (1969).
"Stereochemistry of nucleic acids and their constituents. IV. Allowed and preferred conformations of nucleosides, nucleoside mono-, di-, tri-, tetraphosphates, nucleic acids and polynucleotides"
.
Biopolymers
(en
ingles
)
7
(6): 821?860.
doi
:
10.1002/bip.1969.360070602
.
(require subscricion (
?
))
.
- ↑
R. Langridge, P.J. Gomatos (1963). "The Structure of RNA. Reovirus RNA and transfer RNA have similar three-dimensional structures, which differ from DNA.".
Science
(en
ingles
)
141
(4): 694?698.
PMID
13928677
.
- ↑
Vater A, Klussmann S (January 2015). "Turning mirror-image oligonucleotides into drugs: the evolution of Spiegelmer therapeutics".
Drug Discovery Today
20
(1): 147?155.
PMID
25236655
.
doi
:
10.1016/j.drudis.2014.09.004
.
- ↑
P. Doty, H. Boedtker, J.R. Fresco, R. Haselkorn, M. Litt (1959). "Secondary Structure in Ribonucleic Acids".
Proceedings of the National Academy of Sciences
(en
ingles
)
45
(4): 482?499.
PMID
16590404
.
- ↑
Dardel
F., Kepes F. (2002). Editions de l'Ecole Polytechnique, ed.
Bioinformatique : genomique et post-genomique
. pp. 153?180.
ISBN
978-2730209274
.
- ↑
A.M. Michelson (1958). "Hyperchromicity and nucleic acids.".
Nature
(en
ingles
)
182
(4648): 1502?1503.
PMID
13613306
.
- ↑
Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M, Turner DH; Disney; Childs; Schroeder; Zuker; Turner (2004).
"Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure"
.
Proc. Natl. Acad. Sci. USA
101
(19): 7287?92.
Bibcode
:
2004PNAS..101.7287M
.
PMC
409911
.
PMID
15123812
.
doi
:
10.1073/pnas.0401799101
.
- ↑
Tan ZJ, Chen SJ; Chen (2008).
"Salt dependence of nucleic acid hairpin stability"
.
Biophys. J.
95
(2): 738?52.
Bibcode
:
2008BpJ....95..738T
.
PMC
2440479
.
PMID
18424500
.
doi
:
10.1529/biophysj.108.131524
.
- ↑
K. Hoogsteen (1963). "The crystal and molecular structure of a hydrogen-bonded complex between 1-methylthymine and 9-methyladenine.".
Acta Crystallographica Section A: Foundations of Crystallography
(en
ingles
)
16
: 907?916.
doi
:
10.1107/S0365110X63002437
.
- ↑
H.A. Heus, A. Pardi (1991). "Structural features that give rise to the unusual stability of RNA hairpins containing GNRA loops.".
Science
(en
ingles
)
253
(5016): 191?194.
PMID
1712983
.
- ↑
Staple D.W., Butcher S.E. (2005).
"Pseudoknots: RNA Structures with Diverse Functions."
.
PLoS Biology
(en
ingles
)
3
(6): e213.
PMID
15941360
. Arquivado dende
o orixinal
o 24 de setembro de 2019
. Consultado o 03 de abril de 2015
.
Arquivado
24 de setembro de 2019 en
Wayback Machine
.
- ↑
Costa M., Michel F. (1995).
"Frequent use of the same tertiary motif by self-folding RNAs"
.
EMBO Journal
(en
ingles
)
14
: 1276?1285.
PMID
7720718
.
- ↑
H.R. Drew, R.M. Wing, T. Tanako, C Broka, S Tanaka, K Itakura, R.E. Dickerson (1981).
"Structure of a B-DNA dodecamer: conformation and dynamics."
.
Proc. Natl. Acad. Sci. USA
(en
ingles
)
78
(4): 2179?2183.
PMID
6941276
.
- ↑
Peter S. Klosterman, Sapan A., Shah Thomas A., Steitz (1999).
"Crystal structures of two plasmid copy control related RNA duplexes: An 18 base pair duplex at 1.20 A resolution and a 19 base pair duplex at 1.55 A resolution."
.
Biochemistry
(en
ingles
)
38
(45): 14784?14792.
PMID
10555960
.
doi
:
10.1021/bi9912793
.
- ↑
J.M. Rosenberg, N.C. Seeman, J.J. Kim, F.L. Suddath, H.B. Nicholas, A. Rich (1973).
"Double helix at atomic resolution."
.
Nature
(en
ingles
)
243
(5403): 150?154.
PMID
4706285
. Consultado o 6 de novembro de 2009
.
- ↑
R.O. Day, N.C. Seeman, J.M. Rosenberg, A. Rich (1973). "A Crystalline Fragment of the Double Helix: The Structure of the Dinucleoside Phosphate Guanylyl-3',5'-Cytidine.".
Proc. Natl. Acad. Sci. USA
(en
ingles
)
70
(3): 849?853.
JSTOR
62373
.
PMID
4514996
.
- ↑
Alexander Rich, David R. Davies (1956). "A new two stranded helical structure: Polyadenylic acid and polyuridylic acid".
J. Am. Chem. Soc.
(en
ingles
)
78
(14): 3548?3549.
doi
:
10.1021/ja01595a086
.
- ↑
D.E. Draper (1995).
"Protein-RNA recognition"
.
Annu. Rev. Biochem.
(en
ingles
)
64
: 593?620.
PMID
7574494
.
- ↑
Nudler E, Gottesman ME; Gottesman (2002). "Transcription termination and anti-termination in E. coli".
Genes to Cells
7
(8): 755?68.
PMID
12167155
.
doi
:
10.1046/j.1365-2443.2002.00563.x
.
- ↑
Jeffrey L Hansen, Alexander M Long, Steve C Schultz; Long; Schultz (1997). "Structure of the RNA-dependent RNA polymerase of poliovirus".
Structure
5
(8): 1109?22.
PMID
9309225
.
doi
:
10.1016/S0969-2126(97)00261-X
.
- ↑
Ahlquist P (2002). "RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing".
Science
296
(5571): 1270?73.
Bibcode
:
2002Sci...296.1270A
.
PMID
12016304
.
doi
:
10.1126/science.1069132
.
- ↑
Dreyfus M., Regnier P. (2002).
"The poly(A) tail of mRNAs: bodyguard in eukaryotes, scavenger in bacteria"
.
Cell
(en
ingles
)
111
: 611?613.
PMID
12464173
.
- ↑
Cohn W.E. (1960).
"Pseudouridine, a carbon-carbon linked ribonucleoside in ribonucleic acids: isolation, structure, and chemical characteristics"
(PDF)
.
J. Biol. Chem.
(en
ingles
)
235
: 1488?1498.
PMID
13811056
. Arquivado dende
o orixinal
(PDF)
o 24 de setembro de 2019
. Consultado o 03 de abril de 2015
.
Arquivado
24 de setembro de 2019 en
Wayback Machine
.
- ↑
Kowalak J.A., Dalluge J.J., McCloskey J.A., Stetter K.O. (1994). "The role of posttranscriptional modification in stabilization of transfer RNA from hyperthermophiles.".
Biochemistry
(en
ingles
)
28
: 7869?7876.
PMID
7516708
.
- ↑
42,0
42,1
42,2
Cooper GC, Hausman RE (2004).
The Cell: A Molecular Approach
(3rd ed.). Sinauer. pp. 261?76, 297, 339?44.
ISBN
0-87893-214-3
.
OCLC
174924833
.
- ↑
Mattick JS, Gagen MJ; Gagen (1 September 2001).
"The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms"
.
Mol. Biol. Evol.
18
(9): 1611?30.
PMID
11504843
.
doi
:
10.1093/oxfordjournals.molbev.a003951
.
- ↑
Mattick, JS (2001).
"Noncoding RNAs: the architects of eukaryotic complexity"
.
EMBO Reports
2
(11): 986?91.
PMC
1084129
.
PMID
11713189
.
doi
:
10.1093/embo-reports/kve230
.
- ↑
Mattick JS (October 2003).
"Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms"
(PDF)
.
BioEssays : News and Reviews in Molecular, Cellular and Developmental Biology
25
(10): 930?9.
PMID
14505360
.
doi
:
10.1002/bies.10332
. Arquivado dende
o orixinal
(PDF)
o 06 de marzo de 2009
. Consultado o 19 de marzo de 2015
.
Arquivado
06 de marzo de 2009 en
Wayback Machine
.
- ↑
Mattick JS (October 2004).
"The hidden genetic program of complex organisms"
.
Scientific American
291
(4): 60?7.
PMID
15487671
.
doi
:
10.1038/scientificamerican1004-60
.
- ↑
47,0
47,1
47,2
Wirta W (2006).
Mining the transcriptome ? methods and applications
. Stockholm: School of Biotechnology, Royal Institute of Technology.
ISBN
91-7178-436-5
.
OCLC
185406288
.
- ↑
Rossi JJ (2004). "Ribozyme diagnostics comes of age".
Chemistry & Biology
11
(7): 894?95.
PMID
15271347
.
doi
:
10.1016/j.chembiol.2004.07.002
.
- ↑
Gueneau de Novoa P, Williams KP; Williams (2004).
"The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts"
.
Nucleic Acids Res.
32
(Database issue): D104?8.
PMC
308836
.
PMID
14681369
.
doi
:
10.1093/nar/gkh102
.
- ↑
Wu L, Belasco JG; Belasco (January 2008). "Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs".
Mol. Cell
29
(1): 1?7.
PMID
18206964
.
doi
:
10.1016/j.molcel.2007.12.010
.
- ↑
Matzke MA, Matzke AJM; Matzke (2004).
"Planting the seeds of a new paradigm"
.
PLoS Biology
2
(5): e133.
PMC
406394
.
PMID
15138502
.
doi
:
10.1371/journal.pbio.0020133
.
- ↑
Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert J, Bartel DP, Crete P; Vaucheret; Rajagopalan; Lepers; Gasciolli; Mallory; Hilbert; Bartel; Crete (2004). "Endogenous
trans
-acting siRNAs regulate the accumulation of
Arabidopsis
mRNAs".
Molecular Cell
16
(1): 69?79.
PMID
15469823
.
doi
:
10.1016/j.molcel.2004.09.028
.
- ↑
Watanabe T; Totoki Y; Toyoda A; et al. (May 2008). "Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes".
Nature
453
(7194): 539?43.
Bibcode
:
2008Natur.453..539W
.
PMID
18404146
.
doi
:
10.1038/nature06908
.
- ↑
Sontheimer EJ, Carthew RW; Carthew (July 2005). "Silence from within: endogenous siRNAs and miRNAs".
Cell
122
(1): 9?12.
PMID
16009127
.
doi
:
10.1016/j.cell.2005.06.030
.
- ↑
Doran G (2007).
"RNAi ? Is one suffix sufficient?"
.
Journal of RNAi and Gene Silencing
3
(1): 217?19. Arquivado dende
o orixinal
o 16 de xullo de 2007
. Consultado o 19 de marzo de 2015
.
Arquivado
16 de xullo de 2007 en
Wayback Machine
.
- ↑
Pushparaj PN, Aarthi JJ, Kumar SD, Manikandan J; Aarthi; Kumar; Manikandan (2008).
"RNAi and RNAa ? The Yin and Yang of RNAome"
.
Bioinformation
2
(6): 235?7.
PMC
2258431
.
PMID
18317570
.
doi
:
10.6026/97320630002235
.
- ↑
Horwich MD, Li C Matranga C, Vagin V, Farley G, Wang P, Zamore PD; Li; Matranga; Vagin; Farley; Wang; Zamore (2007). "The
Drosophila
RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC".
Current Biology
17
(14): 1265?72.
PMID
17604629
.
doi
:
10.1016/j.cub.2007.06.030
.
- ↑
Girard A, Sachidanandam R, Hannon GJ, Carmell MA; Sachidanandam; Hannon; Carmell (2006). "A germline-specific class of small RNAs binds mammalian Piwi proteins".
Nature
442
(7099): 199?202.
Bibcode
:
2006Natur.442..199G
.
PMID
16751776
.
doi
:
10.1038/nature04917
.
- ↑
Horvath P, Barrangou R; Barrangou (2010).
"CRISPR/Cas, the Immune System of Bacteria and Archaea"
.
Science
327
(5962): 167?70.
Bibcode
:
2010Sci...327..167H
.
PMID
20056882
.
doi
:
10.1126/science.1179555
.
- ↑
Wagner EG, Altuvia S, Romby P; Altuvia; Romby (2002). "Antisense RNAs in bacteria and their genetic elements".
Adv Genet
. Advances in Genetics
46
: 361?98.
ISBN
9780120176465
.
PMID
11931231
.
doi
:
10.1016/S0065-2660(02)46013-0
.
- ↑
Gilbert SF (2003).
Developmental Biology
(7th ed.). Sinauer. pp.
101
?3.
ISBN
0-87893-258-5
.
OL
8127135M
.
- ↑
Amaral PP, Mattick JS; Mattick (October 2008). "Noncoding RNA in development".
Mammalian genome : official journal of the International Mammalian Genome Society
19
(7?8): 454?92.
PMID
18839252
.
doi
:
10.1007/s00335-008-9136-7
.
- ↑
Heard E, Mongelard F, Arnaud D, Chureau C, Vourc'h C, Avner P; Mongelard; Arnaud; Chureau; Vourc'h; Avner (1999).
"Human
XIST
yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells"
.
Proc. Natl. Acad. Sci. USA
96
(12): 6841?46.
Bibcode
:
1999PNAS...96.6841H
.
PMC
22003
.
PMID
10359800
.
doi
:
10.1073/pnas.96.12.6841
.
- ↑
Batey RT (2006). "Structures of regulatory elements in mRNAs".
Curr. Opin. Struct. Biol.
16
(3): 299?306.
PMID
16707260
.
doi
:
10.1016/j.sbi.2006.05.001
.
- ↑
Scotto L, Assoian RK; Assoian (June 1993).
"A GC-rich domain with bifunctional effects on mRNA and protein levels: implications for control of transforming growth factor beta 1 expression"
.
Mol. Cell. Biol.
13
(6): 3588?97.
PMC
359828
.
PMID
8497272
. Arquivado dende
o orixinal
o 24 de setembro de 2019
. Consultado o 19 de marzo de 2015
.
- ↑
Steitz TA, Steitz JA; Steitz (1993).
"A general two-metal-ion mechanism for catalytic RNA"
.
Proc. Natl. Acad. Sci. U.S.A.
90
(14): 6498?502.
Bibcode
:
1993PNAS...90.6498S
.
PMC
46959
.
PMID
8341661
.
doi
:
10.1073/pnas.90.14.6498
.
- ↑
Xie J, Zhang M, Zhou T, Hua X, Tang L, Wu W; Zhang; Zhou; Hua; Tang; Wu (2007).
"Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs"
.
Nucleic Acids Res.
35
(Database issue): D183?7.
PMC
1669756
.
PMID
17099227
.
doi
:
10.1093/nar/gkl873
.
- ↑
Omer AD, Ziesche S, Decatur WA, Fournier MJ, Dennis PP; Ziesche; Decatur; Fournier; Dennis (2003). "RNA-modifying machines in archaea".
Molecular Microbiology
48
(3): 617?29.
PMID
12694609
.
doi
:
10.1046/j.1365-2958.2003.03483.x
.
- ↑
Cavaille J, Nicoloso M, Bachellerie JP; Nicoloso; Bachellerie (1996). "Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides".
Nature
383
(6602): 732?5.
Bibcode
:
1996Natur.383..732C
.
PMID
8878486
.
doi
:
10.1038/383732a0
.
- ↑
Kiss-Laszlo Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M, Kiss T; Henry; Bachellerie; Caizergues-Ferrer; Kiss (1996). "Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs".
Cell
85
(7): 1077?88.
PMID
8674114
.
doi
:
10.1016/S0092-8674(00)81308-2
.
- ↑
Shippen-Lentz D., Blackburn E.H. (1990). "Functional evidence for an RNA template in telomerase.".
Science
(en
ingles
)
247
: 546?552.
PMID
1689074
.
- ↑
Huarte M., Jacks T., Rinn J.L. (2010). "A Large Intergenic Noncoding RNA Induced by p53 Mediates Global Gene Repression in the p53 Response".
Cell
(en
ingles
)
142
: 409?419.
PMID
20673990
.
- ↑
Kruger K., Grabowski P.J., Zaug A.J., Sands J., Gottschling D.E., Cech T.R. (1982). "Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena.".
Cell
(en
ingles
)
31
: 147?157.
PMID
6297745
.
- ↑
Guerrier-Takada C., Gardiner K., Marsh T., Pace N., Altman S. (1983). "The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme.".
Cell
(en
ingles
)
35
: 849?857.
PMID
6197186
.
- ↑
Forster A.C., Davies C., Hutchins C.J., Symons R.H. (1990). "Characterization of self-cleavage of viroid and virusoid RNAs.".
Methods in Enzymology
(en
ingles
)
181
: 583?607.
PMID
2199768
.
- ↑
Cech T.R. (2000). "Structural biology. The ribosome is a ribozyme.".
Science
(en
ingles
)
289
: 878?879.
PMID
10960319
.
- ↑
S. Valadkhan, A. Mohammadi, Y. Jaladat et S. Geisler. Protein-free small nuclear RNAs catalyze a two-step splicing reaction. Proc. Natl. Acad. Sci. USA, vol. 106,? 2009, p. 11901-11906.
PMID 19549866
- ↑
Barrick J.E., Corbino K.A., Winkler W.C., Nahvi A., Mandal M., Collins J., Lee M., Roth A., Sudarsan N., Jona I., Wickiser J.K., Breaker R.R. (2004). "New RNA motifs suggest an expanded scope for riboswitches in bacterial genetic control.".
Proc. Natl. Acad. Sci. USA
(en
ingles
)
101
: 6421?6426.
PMID
15096624
.
- ↑
Ellington A.D., Szostak J.W. (1990). "In vitro selection of RNA molecules that bind specific ligands.".
Nature
(en
ingles
)
346
: 818?822.
PMID
1697402
.
Tuerk C., Gold L. (1990). "Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase.".
Science
(en
ingles
)
249
: 505?510.
PMID
2200121
.
- ↑
Daros JA, Elena SF, Flores R; Elena; Flores (2006).
"Viroids: an Ariadne's thread into the RNA labyrinth"
.
EMBO Rep.
7
(6): 593?8.
PMC
1479586
.
PMID
16741503
.
doi
:
10.1038/sj.embor.7400706
.
- ↑
Kalendar R, Vicient CM, Peleg O, Anamthawat-Jonsson K, Bolshoy A, Schulman AH; Vicient; Peleg; Anamthawat-Jonsson; Bolshoy; Schulman (2004).
"Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes"
.
Genetics
166
(3): 1437?50.
PMC
1470764
.
PMID
15082561
.
doi
:
10.1534/genetics.166.3.1437
.
- ↑
Podlevsky JD, Bley CJ, Omana RV, Qi X, Chen JJ; Bley; Omana; Qi; Chen (2008).
"The telomerase database"
.
Nucleic Acids Res.
36
(Database issue): D339?43.
PMC
2238860
.
PMID
18073191
.
doi
:
10.1093/nar/gkm700
.
- ↑
Blevins, T.; Rajeswaran, R.; Shivaprasad, PV.; Beknazariants, D.; Si-Ammour, A.; Park, HS.; Vazquez, F.; Robertson, D.; Meins, F. (2006). "Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing.".
Nucleic Acids Res
34
(21): 6233?46.
PMID
17090584
.
doi
:
10.1093/nar/gkl886
.
- ↑
Jana S, Chakraborty C, Nandi S, Deb JK; Chakraborty; Nandi; Deb (2004). "RNA interference: potential therapeutic targets".
Appl. Microbiol. Biotechnol.
65
(6): 649?57.
PMID
15372214
.
doi
:
10.1007/s00253-004-1732-1
.
- ↑
Schultz U, Kaspers B, Staeheli P; Kaspers; Staeheli (2004). "The interferon system of non-mammalian vertebrates".
Dev. Comp. Immunol.
28
(5): 499?508.
PMID
15062646
.
doi
:
10.1016/j.dci.2003.09.009
.
- ↑
Whitehead, K. A.; Dahlman, J. E.; Langer, R. S.; Anderson, D. G. (2011). "Silencing or Stimulation? SiRNA Delivery and the Immune System". Annual Review of Chemical and Biomolecular Engineering 2: 77?96. doi:10.1146/annurev-chembioeng-061010-114133.
PMID 22432611
.
- ↑
Fire A., Xu S., Montgomery M., Kostas S., Driver S., Mello C. (1998). "Potent and specific genetic interference by double-stranded RNA in
Caenorhabditis elegans
.".
Nature
(en
ingles
)
391
: 806?811.
- ↑
Helene Claude (2002). "Les promesses de l'ARN therapeutique = Genetic interference by RNA.".
Le Concours Medical
124
: 2550?2552.
- ↑
Dahm R (2005). "Friedrich Miescher and the discovery of DNA".
Developmental Biology
278
(2): 274?88.
PMID
15680349
.
doi
:
10.1016/j.ydbio.2004.11.028
.
- ↑
Caspersson T, Schultz J; Schultz (1939). "Pentose nucleotides in the cytoplasm of growing tissues".
Nature
143
(3623): 602?3.
Bibcode
:
1939Natur.143..602C
.
doi
:
10.1038/143602c0
.
- ↑
Ochoa S (1959).
"Enzymatic synthesis of ribonucleic acid"
(PDF)
.
Nobel Lecture
.
- ↑
Rich A, Davies, D; Davies (1956). "A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid".
Journal of the American Chemical Society
78
(14): 3548.
doi
:
10.1021/ja01595a086
.
- ↑
Holley RW; et al. (1965). "Structure of a ribonucleic acid".
Science
147
(3664): 1462?65.
Bibcode
:
1965Sci...147.1462H
.
PMID
14263761
.
doi
:
10.1126/science.147.3664.1462
.
- ↑
Siebert S (2006).
"Common sequence structure properties and stable regions in RNA secondary structures"
(PDF)
.
Dissertation, Albert-Ludwigs-Universitat, Freiburg im Breisgau
. p. 1. Arquivado dende
o orixinal
(PDF)
o 09 de marzo de 2012
. Consultado o 19 de marzo de 2015
.
- ↑
Szathmary E (1999). "The origin of the genetic code: amino acids as cofactors in an RNA world".
Trends Genet.
15
(6): 223?9.
PMID
10354582
.
doi
:
10.1016/S0168-9525(99)01730-8
.
- ↑
Fiers W; et al. (1976). "Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene".
Nature
260
(5551): 500?7.
Bibcode
:
1976Natur.260..500F
.
PMID
1264203
.
doi
:
10.1038/260500a0
.
- ↑
Napoli C, Lemieux C, Jorgensen R; Lemieux; Jorgensen (1990).
"Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans"
.
Plant Cell
2
(4): 279?89.
PMC
159885
.
PMID
12354959
.
doi
:
10.1105/tpc.2.4.279
.
- ↑
Dafny-Yelin M, Chung SM, Frankman EL, Tzfira T; Chung; Frankman; Tzfira (December 2007).
"pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants"
.
Plant Physiol.
145
(4): 1272?81.
PMC
2151715
.
PMID
17766396
.
doi
:
10.1104/pp.107.106062
.
- ↑
Ruvkun G (2001). "Glimpses of a tiny RNA world".
Science
294
(5543): 797?99.
PMID
11679654
.
doi
:
10.1126/science.1066315
.
- ↑
Fichou Y, Ferec C; Ferec (2006).
"The potential of oligonucleotides for therapeutic applications"
.
Trends in Biotechnology
24
(12): 563?70.
PMID
17045686
.
doi
:
10.1016/j.tibtech.2006.10.003
.
- ↑
Marlaire, Ruth (3 March 2015).
"NASA Ames Reproduces the Building Blocks of Life in Laboratory"
.
NASA
. Arquivado dende
o orixinal
o 05 de marzo de 2015
. Consultado o
5 March
2015
.