 Animacion de un
ARN mensajero
.
Animacion de un
ARN mensajero
.
El
acido ribonucleico
o
ARN
es un
acido nucleico
formado por una cadena de
ribonucleotidos
.
[
1
]
[
2
]
Esta presente tanto en las
celulas
procariotas
como en las
eucariotas
, y es el unico material genetico de ciertos virus (los
virus ARN
).
El ARN se puede definir como una
molecula
formada por una cadena simple de ribonucleotidos, cada uno de ellos formado por
ribosa
, un
fosfato
y una de las cuatro
bases nitrogenadas
(
adenina
,
guanina
,
citosina
y
uracilo
).
[
3
]
El ARN celular es lineal y monocatenario (de una sola cadena), pero en el genoma de algunos virus es de doble hebra.
[
4
]
En los organismos celulares desempena diversas funciones. Es la
molecula
que dirige las etapas intermedias de la
sintesis proteica
; el
ADN
no puede actuar solo, y se vale del ARN para transferir esta informacion vital durante la sintesis de proteinas (produccion de las proteinas que necesita la celula para sus actividades y su desarrollo).
[
5
]
Varios tipos de ARN regulan la
expresion genica
, mientras que otros tienen actividad
catalitica
. El ARN es, pues, mucho mas versatil que el ADN.
Descubrimiento e historia
[
editar
]
Los
acidos nucleicos
fueron descubiertos en 1867 por
Friedrich Miescher
, que los llamo
nucleina
, ya que los aislo del
nucleo celular
.
[
6
]
Mas tarde, se comprobo que las celulas procariotas, que carecen de nucleo, tambien contenian acidos nucleicos. El papel del ARN en la sintesis de proteinas fue sospechado en 1939.
[
7
]
Severo Ochoa
gano el
Premio Nobel de Medicina
en 1959 tras descubrir como se sintetizaba el ARN.
[
8
]
En 1965
Robert W. Holley
hallo la secuencia de 77 nucleotidos de un ARN de transferencia de una
levadura
,
[
9
]
con lo que obtuvo el Premio Nobel de Medicina en 1968. En 1967,
Carl Woese
comprobo las propiedades
cataliticas
de algunos ARN y sugirio que las primeras formas de vida usaron ARN como portador de la informacion genetica tanto como catalizador de sus reacciones
metabolicas
(
hipotesis del mundo de ARN
).
[
10
]
[
11
]
En 1976,
Walter Fiers
y sus colaboradores determinaron la secuencia completa del ARN del
genoma
de un
virus ARN
(
bacteriofago
MS2
).
[
12
]
En 1990 se descubrio en
Petunia
que genes introducidos pueden silenciar genes similares de la misma planta, lo que condujo al descubrimiento del
ARN interferente
.
[
13
]
[
14
]
Aproximadamente al mismo tiempo se hallaron los
micro-ARN
, pequenas moleculas de 22 nucleotidos que tenian algun papel en el
desarrollo
de
Caenorhabditis elegans
.
[
15
]
El descubrimiento de ARN que regulan la
expresion genica
ha permitido el desarrollo de medicamentos hechos de ARN, como los
ARN pequenos de interferencia
que silencian genes.
[
16
]
En el ano 2023 se tiene comprobado que las moleculas de ARN fueron la primera forma de
vida
propiamente dicha en habitar el planeta Tierra (
Hipotesis del mundo de ARN
).
Bioquimica del ARN
[
editar
]
 Estructura quimica del ARN.
Estructura quimica del ARN.
 Comparacion entre
ARN
y
ADN
.
Comparacion entre
ARN
y
ADN
.
Como el ADN, el ARN esta formado por una cadena de
monomeros
repetitivos llamados nucleotidos. Los nucleotidos se unen uno tras otro mediante enlaces
fosfodiester
cargados negativamente.
Cada nucleotido esta formado por tres componentes:
- Un
monosacarido
de cinco
carbonos
(
pentosa
) llamada
β-D-ribofuranosa
.
- Un grupo
fosfato
- Una
base nitrogenada
, que puede ser
- Adenina (A)
- Citosina (C)
- Guanina (G)
- Uracilo (U)
Comparacion entre el ARN y el ADN
|
ARN
|
ADN
|
| Pentosa
|
Ribosa
|
Desoxirribosa
|
| Purinas
|
Adenina y Guanina
|
Adenina y Guanina
|
| Pirimidinas
|
Citosina y
Uracilo
|
Citosina y
Timina
|
Los carbonos de la ribosa se numeran de 1' a 5' en sentido horario. La
base nitrogenada
se une al carbono 1'; el grupo fosfato se une al carbono 5' y al carbono 3' de la ribosa del siguiente nucleotido. El pico tiene una carga negativa a
pH
fisiologico lo que confiere al ARN caracter
polianionico
. Las bases
puricas
(adenina y guanina) pueden formar
puentes de hidrogeno
con las
pirimidinicas
(uracilo y citosina) segun el esquema C=G y A=U.
[
17
]
Ademas, son posibles otras interacciones, como el
apilamiento de bases
[
18
]
o
tetrabucles
con apareamientos G=A.
[
17
]
Muchos ARN contienen ademas de los nucleotidos habituales, nucleotidos modificados, que se originan por transformacion de los nucleotidos tipicos; son caracteristicos de los
ARN de transferencia
(ARNt) y el
ARN ribosomico
(ARNr); tambien se encuentran nucleotidos
metilados
en el
ARN mensajero
eucariotico.
[
19
]
Apareamiento doble
[
editar
]
 Apareamiento entre guanina y uracilo.
Apareamiento entre guanina y uracilo.
La interaccion por puentes de hidrogeno descrita por Watson y Crick
[
20
]
forma pares de bases entre una purina y una pirimidina. A este patron se le conoce como
apareamiento Watson y Crick
. En este, la adenina se aparea con el uracilo (timina, en ADN) y la citosina con la guanina. Sin embargo, en el ARN se presentan muchas otras formas de apareamiento, de las cuales la mas ubicua es el
apareamiento wobble
(tambien
apareamiento por balanceo o apareamiento titubeante
) para la pareja G-U. Este fue propuesto por primera vez por Crick para explicar el apareamiento codon-anticodon en los ARNt y ha sido confirmado en casi todas las clases de ARN en los tres dominios filogeneticos.
[
21
]
Estructura
[
editar
]
Estructura primaria
[
editar
]
Se refiere a la secuencia lineal de nucleotidos en la molecula de ARN. Los siguientes niveles estructurales (estructura secundaria, terciaria) son consecuencia de la estructura primaria. Ademas, la secuencia misma puede ser informacion funcional; esta puede traducirse para sintetizar proteinas (en el caso del ARNm) o funcionar como region de reconocimiento, region catalitica, entre otras.
Estructura primaria de tRNA
Phe
Estructura secundaria
[
editar
]
 Estructura secundaria de ARN de transferencia: tRNA
Phe
de
S. cerevisiae
.
Estructura secundaria de ARN de transferencia: tRNA
Phe
de
S. cerevisiae
.
El ARN se pliega como resultado de la presencia de regiones cortas con apareamiento intramolecular de bases, es decir, pares de bases formados por secuencias complementarias mas o menos distantes dentro de la misma hebra. La estructura secundaria se refiere, entonces, a las relaciones de apareamiento de bases: ≪El termino ‘estructura secundaria’ denota cualquier patron plano de contactos por apareamiento de bases. Es un concepto topologico y no debe ser confundido con algun tipo de estructura bidimensional≫.
[
22
]
La estructura secundaria puede ser descrita a partir de
motivos estructurales
que se suelen clasificar de la siguiente manera:
Elementos estructurales comunes
[
23
]
| Helice
(tallo, stack)
|
Region con bases apareadas.
|
|
| Bucle
(ciclo, loop)
|
Region incluida en una helice en donde las bases no estan apareadas.
|
|
| Bucle en horquilla
(tallo y bucle, hairpin loop)
|
Estructura en donde regiones cercanas de bases complementarias se aparean, separadas por una region no apareada que permite que la secuencia se doble para formar una helice.
|
|
| Bucle interno
(internal loop)
|
Estructura en donde hay regiones no apareadas en ambos lados de la hebra. Puede ser simetrico o asimetrico.
|
|
| Protuberancia
(buldge)
|
Estructura en donde hay una region no apareada en un solo lado de la hebra.
|
 Representacion grafica 3D del complejo CRISPR/Cas9 Representacion grafica 3D del complejo CRISPR/Cas9
|
| Bucle multiple
(helical junction)
|
Region en donde se juntan multiples helices.
|
|
| Pseudonudo
|
Variacion de bucle en donde solo una parte del bucle si esta apareada. El pseudonudo mas simple consiste de una region libre del ARN apareada con un bucle.
|
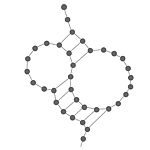
|
Estructura terciaria
[
editar
]
 Estructura terciaria de dos
ARN de transferencia
: tRNA
Phe
y tRNA
Asx
.
Estructura terciaria de dos
ARN de transferencia
: tRNA
Phe
y tRNA
Asx
.
La estructura terciaria es el resultado de las interacciones en el espacio entre los atomos que conforman la molecula. Algunas interacciones de este tipo incluyen el apilamiento de bases y los apareamientos de bases distintos a los propuestos por Watson y Crick, como el apareamiento Hoogsteen, los apareamientos triples y los ziperes de ribosa.
Helice A
[
editar
]
 Helice A de ARN.
Helice A de ARN.
A diferencia del ADN las moleculas de ARN suelen ser de cadena simple y no forman
helices dobles
extensas, no obstante, en las regiones con bases apareadas si forma helices como motivo estructural terciario. Una importante caracteristica estructural del ARN que lo distingue del ADN es la presencia de un grupo
hidroxil
en posicion 2' de la ribosa, que causa que las dobles helices de ARN adopten una conformacion
A
, en vez de la conformacion
B
que es la mas comun en el ADN.
[
24
]
Esta
helice A
tiene un surco mayor muy profundo y estrecho y un surco menor amplio y superficial.
[
25
]
Una segunda consecuencia de la presencia de dicho hidroxilo es que los enlaces fosfodiester del ARN de las regiones en que no se forma helice doble son mas susceptibles de
hidrolisis
quimica que los del ADN; los enlaces fosfodiester del ARN se hidrolizan rapidamente en disolucion
alcalina
, mientras que los enlaces del ADN son estables.
[
26
]
La vida media de las moleculas de ARN es mucho mas corta que las del ADN, de unos minutos en algunos ARN bacterianos o de unos dias en los ARNt humanos.
[
19
]
Biosintesis
[
editar
]
La biosintesis de ARN esta catalizada normalmente por la
enzima
ARN polimerasa
que usa una hebra de
ADN
como molde, proceso conocido con el nombre de
transcripcion
. Por tanto, todos los ARN celulares provienen de copias de
genes
presentes en el
ADN
.
La transcripcion comienza con el reconocimiento por parte de la enzima de un
promotor
, una secuencia caracteristica de nucleotidos en el ADN situada antes del segmento que va a transcribirse; la helice doble del
ADN
es abierta por la actividad
helicasa
de la propia enzima. A continuacion, la
ARN polimerasa
progresa a lo largo de la hebra de
ADN
en sentido 3' → 5', sintetizando una molecula complementaria de ARN; este proceso se conoce como elongacion, y el crecimiento de la molecula de ARN se produce en sentido 5' → 3'. La secuencia de nucleotidos del ADN determina tambien donde acaba la sintesis del ARN, gracias a que posee secuencias caracteristicas que la ARN polimerasa reconoce como senales de terminacion.
[
27
]
Tras la transcripcion, la mayoria de los ARN son modificados por
enzimas
. Por ejemplo, al pre-ARN mensajero eucariota recien transcrito se le anade un nucleotido de guanina modificado (
7-Metilguanosina
) en el extremo 5' por medio de un puente de trifosfato formando un enlace 5'→ 5' unico, tambien conocido como ≪capucha≫ o ≪caperuza≫, y una larga secuencia de nucleotidos de adenina en el extremo 3' (cola poli-A); posteriormente se le eliminan los
intrones
(segmentos no codificantes) en un proceso conocido como
empalme o ayuste
.
En virus, hay tambien varias
ARN polimerasas ARN-dependientes
que usan ARN como molde para la sintesis de nuevas moleculas de ARN. Por ejemplo, varios virus ARN, como los
poliovirus
, usan este tipo de enzimas para replicar su
genoma
.
[
28
]
[
29
]
Clases de ARN
[
editar
]
El
ARN mensajero
(ARNm) es el tipo de ARN que lleva la informacion del ADN a los
ribosomas
, el lugar de la sintesis de proteinas. La secuencia de nucleotidos del ARNm determina la secuencia de
aminoacidos
de la
proteina
.
[
30
]
Por ello, el ARNm es denominado ARN codificante.
No obstante, muchos ARN no codifican proteinas, y reciben el nombre de
ARN no codificantes
; se originan a partir de
genes
propios (
genes ARN
), o son los intrones rechazados durante el proceso de empalme o ayuste. Son ARN no codificantes el
ARN de transferencia
(ARNt) y el
ARN ribosomico
(ARNr), que son elementos fundamentales en el proceso de
traduccion
, y diversos tipos de ARN reguladores.
[
31
]
Ciertos ARN no codificantes, denominados
ribozimas
, son capaces de
catalizar
reacciones quimicas como cortar y unir otras moleculas de ARN,
[
32
]
o formar
enlaces peptidicos
entre
aminoacidos
en el ribosoma durante la
sintesis de proteinas
.
[
33
]
ARN implicados en la sintesis de proteinas
[
editar
]
 Ribosoma 50S mostrando el ARNr (amarillo), las proteinas (azul) y el
centro activo
, la adenina 2486 (rojo).
Ribosoma 50S mostrando el ARNr (amarillo), las proteinas (azul) y el
centro activo
, la adenina 2486 (rojo).
ARN mensajero
[
editar
]
El
ARN mensajero
(ARNm) es el que lleva la informacion sobre la secuencia de
aminoacidos
de la
proteina
desde el
ADN
, lugar en que esta inscrita, hasta el
ribosoma
, lugar en que se sintetizan las proteinas de la
celula
. Es por lo tanto, una molecula intermediaria entre el ADN y la proteina, y el apelativo de ≪mensajero≫, es del todo descriptivo. En los
eucariotas
, el ARNm se sintetiza en el
nucleoplasma
del
nucleo celular
y donde es procesado antes de acceder al
citosol
, donde se hallan los ribosomas, a traves de los poros de la
envoltura nuclear
.
ARN de transferencia
[
editar
]
Los
ARN de transferencia
(ARNt) son cortos polimeros de unos 80 nucleotidos, que transfiere un aminoacido especifico al
polipeptido
en crecimiento; se unen a lugares especificos del ribosoma durante la traduccion. Tienen un sitio especifico para la fijacion del
aminoacido
(extremo 3') y un
anticodon
formado por un triplete de nucleotidos que se une al
codon
complementario del ARNm mediante puentes de hidrogeno.
[
31
]
Estos ARNt, al igual que otros tipos de ARN, pueden ser modificados post-transcripcionalmente por enzimas. La modificacion de alguna de sus bases es crucial para la descodificacion de ARNm y para mantener la estructura tridimensional del ARNt.
[
34
]
ARN ribosomico
[
editar
]
El
ARN ribosomico
(ARNr) se halla combinado con proteinas para formar los ribosomas, donde representa unas 2/3 partes de los mismos. En procariotas, la subunidad mayor del ribosoma contiene dos moleculas de ARNr y la subunidad menor, una. En los eucariotas, la subunidad mayor contiene tres moleculas de ARNr y la menor, una. En ambos casos, sobre el armazon constituido por los ARNm se asocian proteinas especificas. El ARNr es muy abundante y representa el 80 % del ARN hallado en el
citoplasma
de las celulas eucariotas.
[
35
]
Los ARN ribosomicos son el componente catalitico de los ribosomas; se encargan de crear los enlaces peptidicos entre los aminoacidos del polipeptido en formacion durante la sintesis de proteinas; actuan, pues, como
ribozimas
.
ARN reguladores
[
editar
]
Muchos tipos de ARN regulan la
expresion genica
gracias a que son complementarios de regiones especificas del ARNm o de genes del ADN.
ARN de interferencia
[
editar
]
Los
ARN interferentes
(ARNi) son moleculas de ARN que suprimen la expresion de genes especificos mediante mecanismos conocidos globalmente como
ribointerferencia
o interferencia por ARN. Los ARN interferentes son moleculas pequenas (de 20 a 25 nucleotidos) que se generan por fragmentacion de precursores mas largos. Se pueden clasificar en tres grandes grupos:
[
36
]
Micro-ARN
[
editar
]
Los
micro-ARN
(miARN) son cadenas cortas de 21 o 22 nucleotidos hallados en celulas eucariotas que se generan a partir de precursores especificos codificados en el
genoma
. Al transcribirse, se pliegan en horquillas intramoleculares y luego se unen a enzimas formando un complejo efector que puede bloquear la traduccion del ARNm o acelerar su degradacion comenzando por la eliminacion enzimatica de la cola poli A.
[
37
]
[
38
]
ARN interferente pequeno
[
editar
]
Los ARN interferentes pequenos (ARNip o
siARN
), formados por 20-25 nucleotidos, se producen con frecuencia por rotura de ARN
virales
, pero pueden ser tambien de origen endogeno.
[
39
]
[
40
]
Tras la transcripcion se ensambla en un complejo proteico denominado RISC (
RNA-induced silencing complex
) que identifica el ARNm complementario que es cortado en dos mitades que son degradadas por la maquinaria celular, bloquean asi la expresion del gen.
[
41
]
[
42
]
[
43
]
ARN asociados a Piwi
[
editar
]
Los
ARN asociados a Piwi
[
44
]
son cadenas de 29-30 nucleotidos, propias de
animales
; se generan a partir de precursores largos monocatenarios (formados por una sola cadena), en un proceso que es independiente de
Drosha
y
Dicer
. Estos ARN pequenos se asocian con una subfamilia de las proteinas "Argonauta" denominada proteinas
Piwi
. Son activos las celulas de la
linea germinal
; se cree que son un sistema defensivo contra los
transposones
y que juegan algun papel en la
gametogenesis
.
[
45
]
[
46
]
ARN antisentido
[
editar
]
Un ARN
antisentido
es la hebra complementaria (no codificadora) de un hebra ARNm (codificadora). La mayoria inhiben genes, pero unos pocos activan la transcripcion.
[
47
]
El ARN antisentido se aparea con su ARNm complementario formando una molecula de hebra doble que no puede traducirse y es degradada enzimaticamente.
[
48
]
La introduccion de un
transgen
codificante para un ARNm antisentido es una tecnica usada para bloquear la expresion de un gen de interes. Un mARN antisentido marcado radioactivamente puede usarse para mostrar el nivel de transcripcion de genes en varios tipos de celulas. Algunos tipos estructurales antisentidos son experimentales, ya que se usan como
terapia antisentido
.
ARN largo no codificante
[
editar
]
Muchos
ARN largos no codificantes
(ARNnc largo) regulan la expresion genica en eucariotas;
[
49
]
uno de ellos es el
Xist
que recubre uno de los dos
cromosomas X
en las hembras de los
mamiferos
inactivandolo (
corpusculo de Barr
).
[
50
]
Diversos estudios revelan que es activo a bajos niveles. En determinadas poblaciones celulares, una cuarta parte de los genes que codifican para
proteinas
y el 80 % de los lncRNA detectados en el
genoma humano
estan presentes en una o ninguna copia por celula, ya que existe una restriccion en determinados ARN.
[
51
]
Ribointerruptor
[
editar
]
Un
interruptor molecular o ribointerruptor
es una parte del ARNm (acido ribonucleico mensajero) al cual pueden unirse pequenas moleculas que afectan la actividad del gen.
[
52
]
[
53
]
[
54
]
Por tanto, un ARNm que contenga un interruptor molecular esta directamente implicado en la regulacion de su propia actividad que depende de la presencia o ausencia de la molecula senalizadora. Tales ribointerruptores se hallan en la region no traducida 5' (5'-UTR), situada antes del codon de inicio (AUG), y/o en la region no traducida 3' (3'-UTR), tambien llamada secuencia de arrastre,
[
19
]
situada entre el codon de terminacion (UAG, UAA o UGA) y la cola poli A.
[
54
]
ARN con actividad catalitica
[
editar
]
 Transformacion de
uridina
en
pseudouridina
, una modificacion comun del ARN.
Transformacion de
uridina
en
pseudouridina
, una modificacion comun del ARN.
Ribozimas
[
editar
]
El ARN puede actuar como
biocatalizador
. Ciertos ARN se asocian a proteinas formando
ribonucleoproteinas
y se ha comprobado que es la subunidad de ARN la que lleva a cabo las reacciones cataliticas; estos ARN realizan las reacciones
in vitro
en ausencia de proteina. Se conocen cinco tipos de ribozimas; tres de ellos llevan a cabo reacciones de automodificacion, como eliminacion de
intrones
o autocorte, mientras que los otros (
ribonucleasa P
y ARN ribosomico) actuan sobre substratos distintos.
[
19
]
Asi, la ribonucleasa P corta un ARN precursor en moleculas de ARNt,
[
55
]
mientras que el ARN ribosomico realiza el
enlace peptidico
durante la sintesis proteica ribosomica.
Espliceosoma
[
editar
]
Los
intrones
son separados del pre-ARNm durante el proceso conocido como ≪
ayuste
≫ por los
espliceosomas
, que contienen numerosos
ARN pequenos nucleares
(ARNpn).
[
56
]
En otros casos, los propios intrones actuan como
ribozimas
y se separan a si mismos de los
exones
.
[
57
]
ARN pequeno nucleolar
[
editar
]
Los
ARN pequenos nucleolares
(ARNpno), hallados en el
nucleolo
y en los
cuerpos de Cajal
, dirigen la modificacion de nucleotidos de otros ARN;
[
31
]
el proceso consiste en transformar alguna de las cuatro bases nitrogenadas tipicas (A, C, U, G) en otras. Los ARNpno se asocian con enzimas y los guian apareandose con secuencias especificas del ARN al que modificaran. Los ARNr y los ARNt contienen muchos nucleotidos modificados.
[
58
]
[
59
]
ARN mitocondrial
[
editar
]
La
mitocondrias
tienen su propio aparato de sintesis proteica, que incluye ARNr (en los ribosomas), ARNt y ARNm.
Los
ARN mitocondriales
(ARNmt o mtARN) representan el 4 % del ARN celular total. Son transcritos por una
ARN polimerasa
mitocondrial especifica.
[
19
]
Genomas de ARN
[
editar
]
El ADN es la molecula portadora de la informacion genetica en todos los organismos celulares, pero, al igual que el ADN, el ARN puede guardar informacion genetica. Los
virus ARN
carecen por completo de ADN y su genoma esta formado por ARN, el cual codifica las proteinas del virus, como las de la
capside
y algunos enzimas. Dichos enzimas realizan la
replicacion
del genoma virico. Los
viroides
son otro tipo de
patogenos
que consisten exclusivamente en una molecula de ARN que no codifica ninguna proteina y que es replicado por la maquinaria de la celula
hospedadora
.
[
60
]
Hipotesis del mundo de ARN
[
editar
]
La hipotesis del mundo de ARN propone que el ARN fue el primer acido nucleico que aparecio en la Tierra precediendo posteriormente al ADN y a su vez estos acidos nucleicos junto con
proteinas
al unirse con
liposomas
formados espontaneamente originarian las primeras celulas. Se basa en la comprobacion de que el ARN puede contener informacion genetica, de un modo analogo a como lo hace el ADN, y que algunos tipos son capaces de llevar a cabo reacciones metabolicas, como autocorte o formacion de enlaces peptidicos.
Durante anos se especulo en que fue primero, el ADN o las enzimas, ya que las enzimas se sintetizan a partir del ADN y la sintesis de ADN es llevada a cabo por enzimas. Si se supone que las primeras formas de vida usaron el ARN tanto para almacenar su informacion genetica como realizar su metabolismo, se supera este escollo. Experimentos con los ribozimas basicos, como el
ARN viral
Q-beta
, han demostrado que las estructuras de ARN autorreplicantes sencillas pueden resistir incluso a fuertes presiones selectivas (como los terminadores de cadena de
quiralidad
opuesta).
[
61
]
Vease tambien
[
editar
]
Referencias
[
editar
]
- ↑
Barioglio, Carlos Fernando (2013).
Diccionario de produccion animal. Edicion
. Editorial Brujas.
ISBN
9789879452561
. Consultado el 1 de octubre de 2019
.
- ↑
Real Academia Espanola.
≪acido ribonucleico≫
.
Diccionario de la lengua espanola
(23.ª edicion).
- ↑
≪acido ribonucleico≫
.
Real Academia Nacional de Medicina
.
- ↑
Bioquimica: libro de texto con aplicaciones clinicas
, pag. 78.
En Google Libros.
- ↑
Prueba de Acceso a la Universidad Para Mayores de 25 Anos. Biologia Prueba Especifica.e-book
. MAD-Eduforma.
ISBN
9788466517775
. Consultado el 1 de octubre de 2019
.
- ↑
Dahm, R. (2005). ≪Friedrich Miescher and the discovery of DNA≫.
Developmental Biology
278
(2): 274-88.
PMID
15680349
.
doi
:
10.1016/j.ydbio.2004.11.028
.
- ↑
- ↑
Ochoa, S. (1959).
≪Enzymatic synthesis of ribonucleic acid≫
.
Nobel Lecture
.
- ↑
Holley, R. W.
et al
(1965). ≪Structure of a ribonucleic acid≫.
Science
147
(1664): 1462-65.
PMID
14263761
.
doi
:
10.1126/science.147.3664.1462
.
- ↑
Siebert, S. (2006).
≪Common sequence structure properties and stable regions in RNA secondary structures≫
.
Dissertation, Albert-Ludwigs-Universitat, Freiburg im Breisgau
. p. 1. Archivado desde
el original
el 9 de marzo de 2012.
- ↑
Szathmary, E. (1999). ≪The origin of the genetic code: amino acids as cofactors in an RNA world≫.
Trends Genet.
15
(6): 223-9.
PMID
10354582
.
doi
:
10.1016/S0168-9525(99)01730-8
.
- ↑
Fiers, W.
et al
(1976). ≪Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene≫.
Nature
260
: 500-7.
PMID
1264203
.
doi
:
10.1038/260500a0
.
- ↑
Napoli, C., Lemieux, C. & Jorgensen, R. (1990).
≪Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans≫
.
Plant Cell
2
(4): 279-89.
PMID
12354959
.
doi
:
10.1105/tpc.2.4.279
.
- ↑
Dafny-Yelin, M., Chung, S.M., Frankman, E. L. & Tzfira, T. (diciembre de 2007).
≪pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants≫
.
Plant Physiol.
145
(4): 1272-81.
PMC
2151715
.
PMID
17766396
.
doi
:
10.1104/pp.107.106062
.
- ↑
Ruvkun, G. (2001). ≪Glimpses of a tiny RNA world≫.
Science
294
(5543): 797-99.
PMID
11679654
.
doi
:
10.1126/science.1066315
.
- ↑
Fichou, Y. & Ferec, C. (2006).
≪The potential of oligonucleotides for therapeutic applications≫
.
Trends in Biotechnology
24
(12): 563-70.
PMID
17045686
.
doi
:
10.1016/j.tibtech.2006.10.003
.
- ↑
a
b
Lee, J. C. & Gutell, R. R. (2004). ≪Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs≫.
J. Mol. Biol.
344
(5): 1225-49.
PMID
15561141
.
doi
:
10.1016/j.jmb.2004.09.072
.
- ↑
Barciszewski, J., Frederic, B. & Clark, C. (1999).
RNA biochemistry and biotechnology
. Springer. pp. 73-87.
ISBN
0792358627
.
OCLC
52403776
.
- ↑
a
b
c
d
e
Devlin, T. M. 2004.
Bioquimica
, 4.ª edicion. Reverte, Barcelona.
ISBN 84-291-7208-4
- ↑
Watson, J. D., & Crick, F. H. (1953). ≪Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid≫.
Nature
171
: 737-738.
- ↑
Brown, T. (2004).
Genomas
. Ed. Medica Paanamericana.
- ↑
Fontana, W. (2002). ≪Modelling “evo-devo” with RNA≫.
BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology
24
(12): 1164-77.
doi
:
10.1002/bies.10190
.
- ↑
Tabla recopilada en
Quinones Olvera, N.
Implicaciones evolutivas de la plasticidad del RNA
.
(Tesis) Universidad Nacional Autonoma de Mexico
- ↑
Salazar, M., Fedoroff, O. Y., Miller, J. M., Ribeiro, N. S., Reid, B. R. (1992). ≪The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution≫.
Biochemistry
. 32 numero=16: 4207-15.
PMID
7682844
.
doi
:
10.1021/bi00067a007
.
- ↑
Hermann, T. & Patel, D. J. (2000). ≪RNA bulges as architectural and recognition motifs≫.
Structure
8
(3): R47?R54.
PMID
10745015
.
doi
:
10.1016/S0969-2126(00)00110-6
.
- ↑
Mikkola, S., Nurmi, K., Yousefi-Salakdeh, E., Stromberg, R. & Lonnberg, H. (1999). ≪The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group≫.
Perkin transactions 2
: 1619-26.
doi
:
10.1039/a903691a
.
- ↑
Nudler, E. & Gottesman, M. E. (2002). ≪Transcription termination and anti-termination in
E. coli
≫.
Genes to Cells
7
: 755-68.
PMID
12167155
.
doi
:
10.1046/j.1365-2443.2002.00563.x
.
- ↑
Hansen, J. L., Long, A. M. & Schultz, S. C. (1997). ≪Structure of the RNA-dependent RNA polymerase of poliovirus≫.
Structure
5
(8): 1109-22.
PMID
9309225
.
doi
:
10.1016/S0969-2126(97)00261-X
.
- ↑
Ahlquist, P. (2002). ≪RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing≫.
Science
296
(5571): 1270-73.
PMID
12016304
.
doi
:
10.1126/science.1069132
.
- ↑
Cooper, G. C., Hausman, R. E. (2004).
The Cell: A Molecular Approach
(3rd edicion). Sinauer. pp. 261-76, 297, 339-44.
ISBN
0-87893-214-3
.
OCLC
174924833 52121379 52359301 56050609
.
- ↑
a
b
c
Wirta, W. (2006).
Mining the transcriptome ? methods and applications
. Stockholm: School of Biotechnology, Royal Institute of Technology.
ISBN
91-7178-436-5
.
OCLC
185406288
.
- ↑
Rossi JJ (2004). ≪Ribozyme diagnostics comes of age≫.
Chemistry & Biology
11
(7): 894-95.
doi
:
10.1016/j.chembiol.2004.07.002
.
- ↑
Nissen, P., Hansen, J., Ban, N., Moore, P. B., Steitz, T. A. (2000). ≪The structural basis of ribosome activity in peptide bond synthesis≫.
Science
289
(5481): 920-30.
PMID
10937990
.
doi
:
10.1126/science.289.5481.920
.
- ↑
Grosjean, H. (2009) Nucleic acids are not boring long polymers of
only four types of nucleotides: a guided tour. DNA and RNA
Modification Enzymes: Structure, Mechanism, Function and
Evolution. Landes Biosciencies, Texas, USA, pp. 1?18.
- ↑
Kampers, T., Friedhoff, P., Biernat, J., Mandelkow, E.-M., Mandelkow, E. (1996). ≪RNA stimulates aggregation of microtubule-associated protein tau into Alzheimer-like paired helical filaments≫.
FEBS Letters
399
: 104D.
PMID
8985176
.
doi
:
10.1016/S0014-5793(96)01386-5
.
- ↑
Grosshans, H. & Filipowicz, W. 2008. Molecular biology: the expanding world of small RNAs.
Nature
, 451(7177):414-6
[1]
- ↑
Wu, L., Belasco, J.G. (enero de 2008). ≪Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs≫.
Mol. Cell
29
(1): 1-7.
PMID
18206964
.
doi
:
10.1016/j.molcel.2007.12.010
.
- ↑
Matzke MA, Matzke AJM (2004). ≪Planting the seeds of a new paradigm≫.
PLoS Biology
2
(5): e133
.
PMID
15138502
.
doi
:
10.1371/journal.pbio.0020133
.
- ↑
Vazquez, F., Vaucheret, H., Rajagopalan, R., Lepers, C., Gasciolli, V., Mallory, A. C., Hilbert, J., Bartel, D. P. & Crete, P. (2004). ≪Endogenous
trans
-acting siRNAs regulate the accumulation of
Arabidopsis
mRNAs≫.
Molecular Cell
16
(1): 69-79.
PMID
15469823
.
doi
:
10.1016/j.molcel.2004.09.028
.
- ↑
Watanabe, T., Totoki, Y., Toyoda, A.,
et al
(mayo de 2008). ≪Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes≫.
Nature
453
(7194): 539-43.
PMID
18404146
.
doi
:
10.1038/nature06908
.
- ↑
Sontheimer, E. J., Carthew, R. W. (julio de 2005). ≪Silence from within: endogenous siRNAs and miRNAs≫.
Cell
122
(1): 9-12.
PMID
16009127
.
doi
:
10.1016/j.cell.2005.06.030
.
- ↑
Doran, G. (2007).
≪RNAi ? Is one suffix sufficient?≫
.
Journal of RNAi and Gene Silencing
3
(1): 217-19. Archivado desde
el original
el 16 de julio de 2007.
- ↑
Pushparaj, P. N., Aarthi, J. J., Kumar, S. D., Manikandan, J. (2008).
≪RNAi and RNAa - The Yin and Yang of RNAome≫
.
Bioinformation
2
(6): 235-7.
PMC
2258431
.
PMID
18317570
.
- ↑
Hartig, J.V., Tomari, Y., Forstemann, K. (2007). ≪piRNAs--the ancient hunters of genome invaders.≫.
Genes Dev.
21
(14).
1707-13
.
[2]
- ↑
Horwich, M. D., Li, C., Matranga, C., Vagin, V., Farley, G., Wang, P, & Zamore, P. D. (2007). ≪The
Drosophila
RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC≫.
Current Biology
17
: 1265-72.
PMID
17604629
.
doi
:
10.1016/j.cub.2007.06.030
.
- ↑
Girard, A., Sachidanandam, R., Hannon, G. J. & Carmell, M. A. (2006). ≪A germline-specific class of small RNAs binds mammalian Piwi proteins≫.
Nature
442
: 199-202.
PMID
16751776
.
doi
:
10.1038/nature04917
.
- ↑
Wagner, E. G., Altuvia, S., Romby, P. (2002). ≪Antisense RNAs in bacteria and their genetic elements≫.
Adv Genet.
46
: 361-98.
PMID
11931231
.
doi
:
10.1016/S0065-2660(02)46013-0
.
- ↑
Gilbert, S. F. (2003).
Developmental Biology
(7th edicion). Sinauer. pp.
101
?3.
ISBN
0878932585
.
OCLC
154656422 154663147 174530692 177000492 177316159 51544170 54743254 59197768 61404850 66754122
.
- ↑
Amaral, P. P., Mattick, J. S. (octubre de 2008). ≪Noncoding RNA in development≫.
Mammalian genome : official journal of the International Mammalian Genome Society
19
: 454.
PMID
18839252
.
doi
:
10.1007/s00335-008-9136-7
.
- ↑
Heard, E., Mongelard, F., Arnaud, D., Chureau, C., Vourc'h, C. & Avner, P. (1999). ≪Human
XIST
yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells≫.
Proc. Natl. Acad. Sci. USA
96
(12): 6841-46.
PMID
10359800
.
doi
:
10.1073/pnas.96.12.6841
.
- ↑
Djebali (2012).
Landscape of transcription in human cells
(489). pp. 101-108
. Consultado el 13 de marzo de 2015
.
- ↑
Tucker, B.J., Breaker, R.R. (2005). ≪Riboswitches as versatile gene control elements≫.
Curr Opin Struct Biol
15
(3): 342-8.
PMID
15919195
.
doi
:
10.1016/j.sbi.2005.05.003
.
- ↑
Vitreschak, A.G., Rodionov, D.A., Mironov, A.A. & Gelfand, M.S. (2004).
≪Riboswitches: the oldest mechanism for the regulation of gene expression?≫
.
Trends Genet
20
(1): 44-50.
PMID
14698618
.
doi
:
10.1016/j.tig.2003.11.008
.
- ↑
a
b
Batey, R.T. (2006). ≪Structures of regulatory elements in mRNAs≫.
Curr Opin Struct Biol
16
(3): 299-306.
PMID
16707260
.
doi
:
10.1016/j.sbi.2006.05.001
.
- ↑
Guerrier-Takada, C., Gardiner, K., Marsh, T., Pace, N. & Altman, S. (1983). ≪The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme≫.
Cell
35
(3 Pt 2): 849-57.
PMID
6197186
.
doi
:
10.1016/0092-8674(83)90117-4
.
- ↑
Berg, J. M., Tymoczko, J. L., Stryer, L. (2002).
Biochemistry
(5th edicion). WH Freeman and Company. pp. 118-19, 781-808.
ISBN
0-7167-4684-0
.
OCLC
179705944 48055706 59502128
.
- ↑
Steitz, T. A., Steitz, J. A. (1993). ≪A general two-metal-ion mechanism for catalytic RNA≫.
Proc. Natl. Acad. Sci. U.S.A.
90
(14): 6498-502.
PMID
8341661
.
doi
:
10.1073/pnas.90.14.6498
.
- ↑
Xie, J., Zhang, M., Zhou, T., Hua, X., Tang, L., Wu, W. (2007). ≪Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs≫.
Nucleic Acids Res.
35
: D183-7.
PMID
17099227
.
doi
:
10.1093/nar/gkl873
.
- ↑
Omer, A. D., Ziesche, S., Decatur, W. A., Fournier, M. J., Dennis, P. P. (2003). ≪RNA-modifying machines in archaea≫.
Molecular Microbiology
48
(3): 617-29.
PMID
12694609
.
doi
:
10.1046/j.1365-2958.2003.03483.x
.
- ↑
Daros, J. A., Elena. S. F., Flores, R. (2006). ≪Viroids: an Ariadne's thread into the RNA labyrinth≫.
EMBO Rep.
7
(6): 593-8.
PMID
16741503
.
doi
:
10.1038/sj.embor.7400706
.
- ↑
Bell, G. 1997.
The Basics of Selection
. Springer, London, 378 pp. ISBN 412 055317
Enlaces externos
[
editar
]